Inno-ADMET
1. Inno-ADMET 概述
ADMET 模块主要用于系统的评价药物的吸收、分布、代谢、排泄和毒性性质,也包括一些理化性质和药物化学性质。众多研究表明早期的 ADMET 性质评价可显著地提高药物研发的成功率。针对不同的需求,系统提供了两个不同的模型来计算成药性性质,分别为基于预训练的 MERT 模型和基于 GNN 子结构可解释性的 MGA 模型。基于自主收集的高质量数据,这两个模型均可实现 17 种物化性质、5 种药物化学性质、6 种类药性规则、21 种 ADME 性质、27 种毒性性质的系统性评价。从模型评估指标的角度来说,MERT 模型更为优秀,根据模型能否提供子结构对预测结果的影响上判断,MGA 模型更具优势,用户可根据自己的需要去选择相应的计算模型。
2. 使用说明
用户只需要四个步骤就可以完成计算:选择方法(默认选择 MERT)-确定输入方式(输入 smiles)-任务命名(可忽略)-提交任务(必点)。
(1) 选择方法
- MERT(Pre-train):为基于预训练的模型,模型的精度更高,预测能力更好,且速度快,计算一个分子只需 2.35ms;
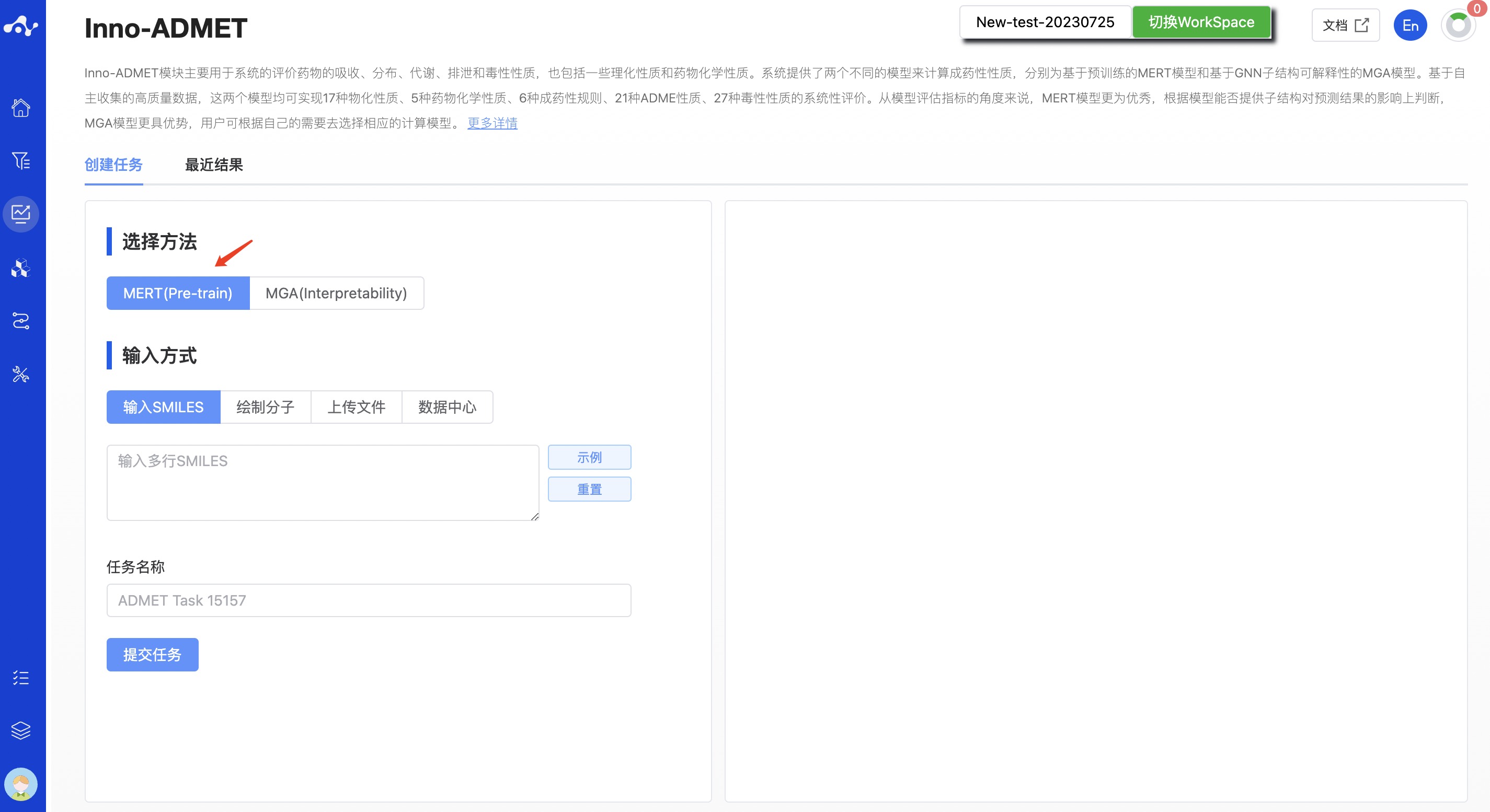
图 1. 创建 Inno-ADMET 任务页面——MERT 模型
- MGA (Interpretability):基于 GNN 子结构可解释性的 MGA 模型,模型预测效果不如 MERT(Pre-train),但该方法能够有效的计算子结构对预测结果的贡献度,有助于挖掘构效关系,在一定程度上可以指导结构优化。选择 MGA 进行计算时,有一个子结构可解释性的开关,由用户决定是否计算子结构可解释性。如打开该开关,系统将会计算子结构对属性的影响,但同时也会增加计算时间,不如 MERT 方法。
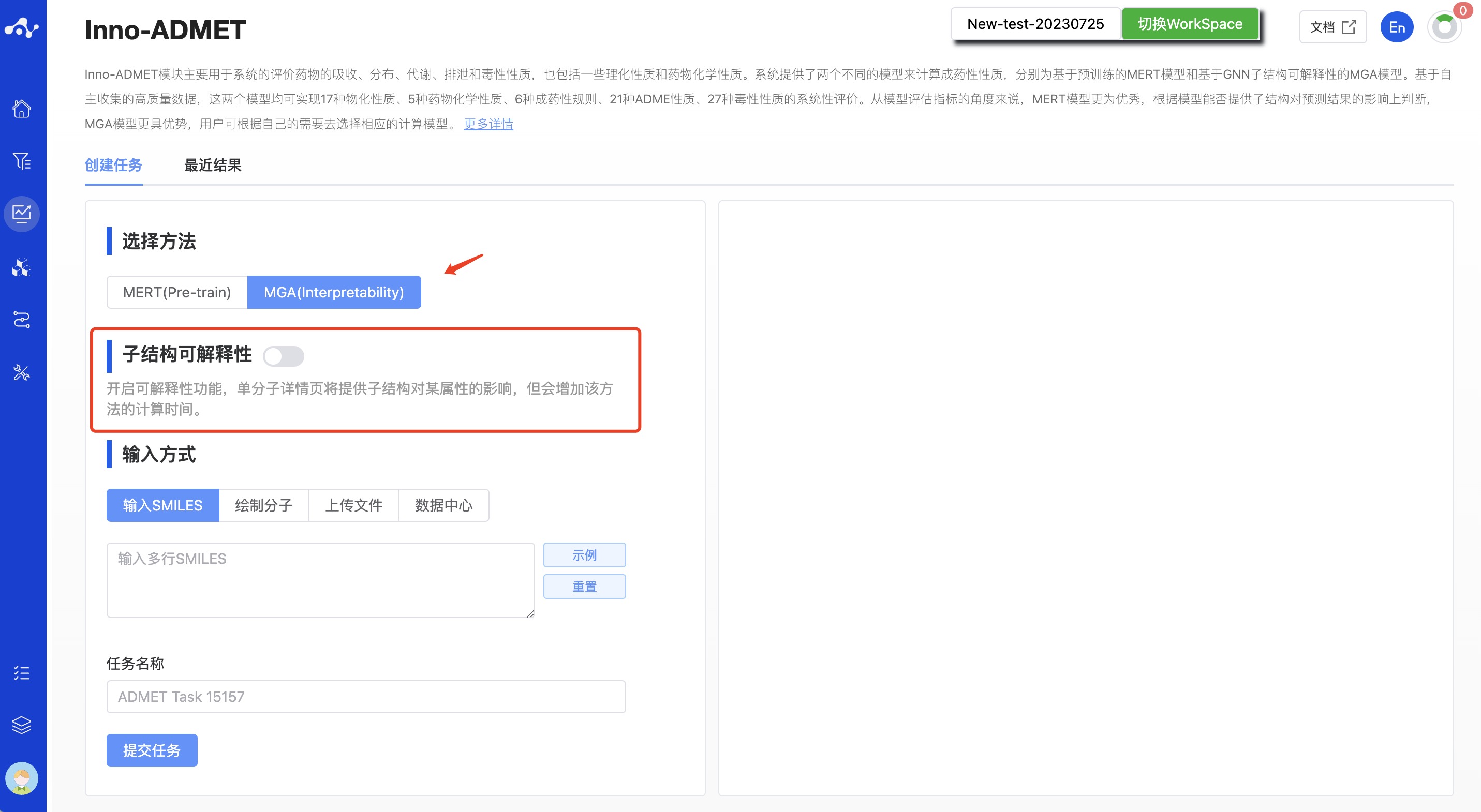
图 2. 创建 Inno-ADMET 任务页面——MGA 模型
(2) 输入方式
平台提供了四种数据输入方式:输入 SMILES、绘制分子、上传文件和数据中心。
- 输入 SMILES
复选框选中“输入 SMILES”,在文本框中输入一个或多个 SMILES 表达式(用换行的方式输入多个 SMILES),该文本框中最多可输入 500 个 SMILES(超出上限可通过上传文件的形式提交任务)。
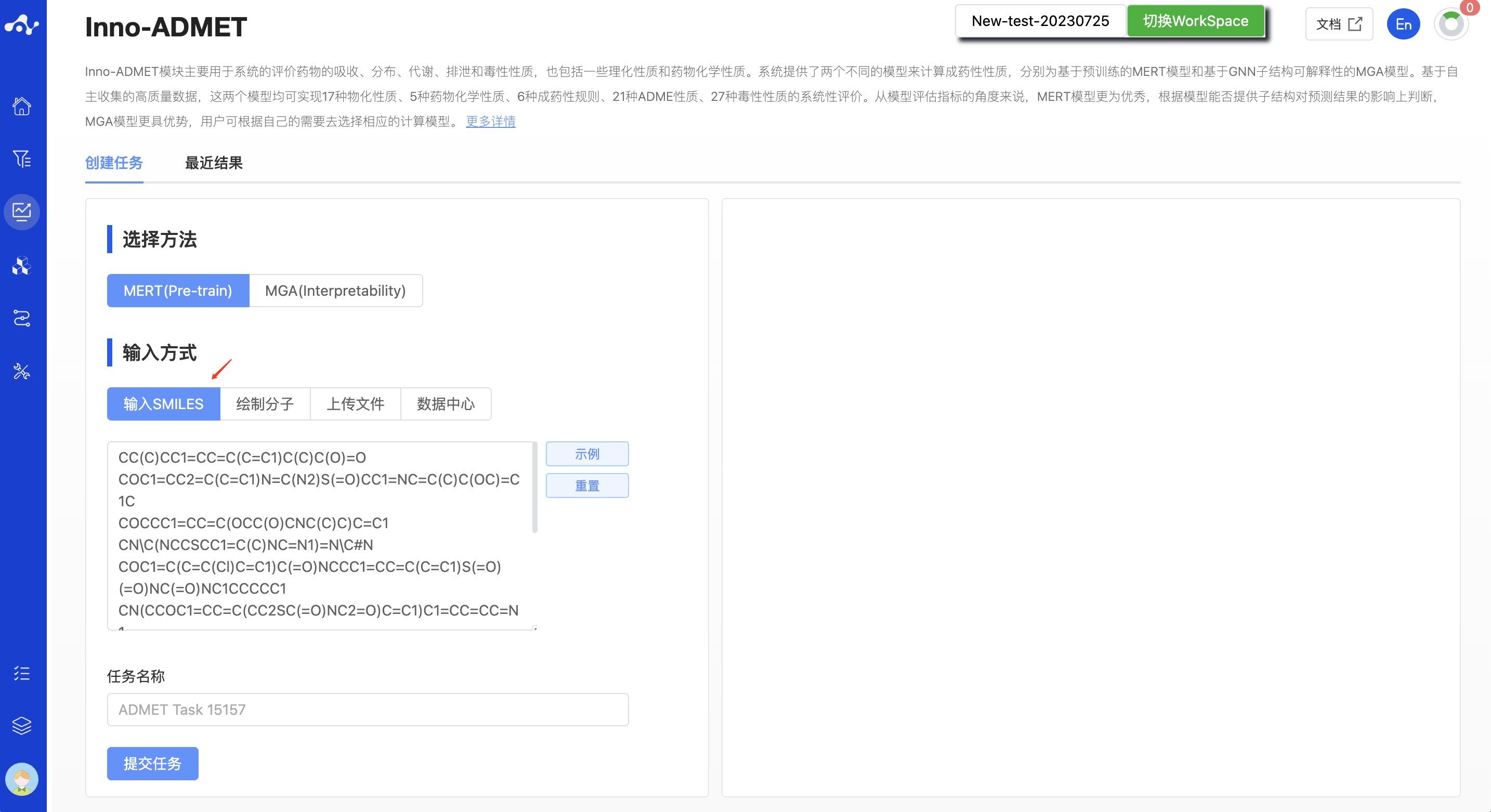
图 3. 输入方式——输入 SMILES
- 绘制分子
复选框选中“绘制分子”,在右边的分子编辑器中画出分子结构,画好以后点击右下角的“确认”后即可。该模式下只支持画一个分子。
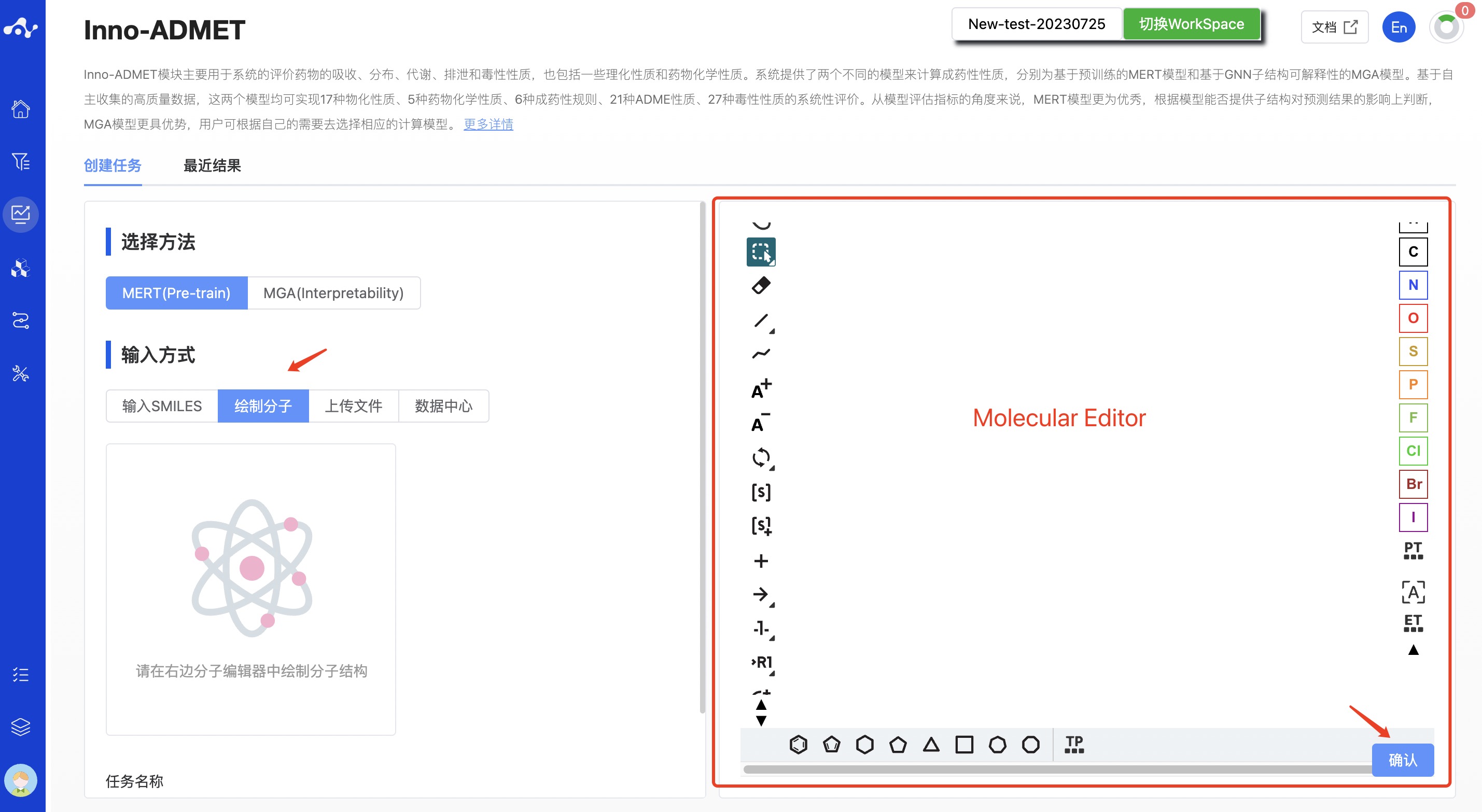
图 4. 输入方式——绘制分子
- 上传文件
复选框选中“上传文件”,通过点击下方按钮选择本地文件即可。选择完文件以后,该文件名称将显示在按钮上,右边将显示文件内容。 关于上传的文件:
当前支持的文件格式: .sdf/.csv;如上传 sdf 文件,则可直接提交任务,如上传 csv 文件,则需指定 smiles 列后方能提交任务。
文件大小不超过 10MB。
- 数据中心
复选框选中“数据中心”,通过点击下方按钮页面出现弹窗,点击文件名称来选择数据中心的数据,点击完之后弹窗消失,即可提交任务。
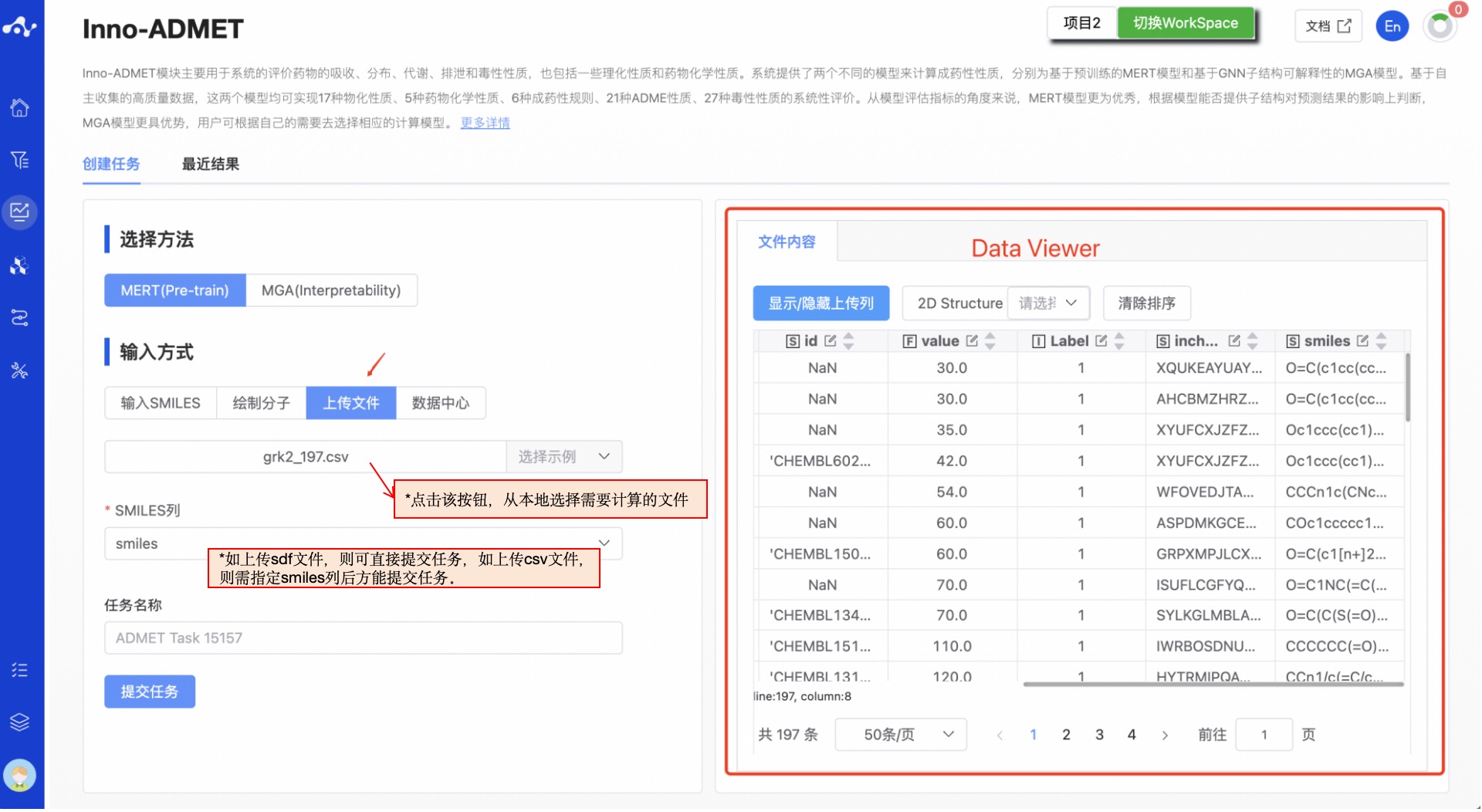
图 5. 输入方式——上传文件/数据中心
(3) 运行进度和结果查看
提交任务后,页面会自动跳入当前页面的“最近结果”子页面中,你可以在该页面查看当前模块的任务运行状态(进度条),也可在右上角的“运行中“下拉框中查看所有模块正在运行的任务。当数据量较大时,系统会分批计算,因此只要有一批数据算完后(整个任务还在运行中),即可点击“结果详情”按钮进入结果页面,查看当前已完成计算的预测结果列表(未完成计算的分子暂不显示),并且可以在当前页面通过刷新来获取最新算完的数据。

图 6. 查看结果
3. 结果分析
结果页面由顶部的 Summary、左侧的筛选区和中部的结果详情区组成。默认状态下结果详情区展示的是结果列表(还可以切换卡片和图表子页面),此时你可以查看预测的所有性质,并对这些性质进行排序、筛选;同时为了方便用户直观的分析数据,我们给预测结果分配了一个颜色。

图 7. 结果页面功能分布
(1) 颜色定义
由于不同的性质有不同的建议取值区间,因此我们通过颜色对预测结果做了直观的评估。颜色设置的规则如下:
- 针对预测结果为 0-1 之间的性质:
P≤0.5,数值的底色为绿色,代表预测的化合物不属于该类别的化合物;
05<P<0.7,数值的底色为黄色,代表预测化合物有可能属于该类化合物;
P≥0.7,数值的底色为红色,代表预测的化合物很有可能属于该类化合物。
- 针对有最优区间的性质,如:
Fsp3 的最优取值为 Fsp3≥0.42,定义两个颜色,符合该阈值的为绿色,不符合的为红色;
Log S 的最优区间为-4~0.5,定义两个颜色,在区间内的为绿色,超出区间的为红色;
QED 的取值有三种定义:Attractive: > 0.67; unattractive: 0.49-0.67; too complex: < 0.34,按照该定义分配红绿黄的颜色即可。
通过以上颜色区分,你可以更直观的了解了解分子的整体评价结果。绿色背景越多,说明分子的 ADMET 性质越好,反之亦然。
(2) 筛选
平台提供了常规筛选和高级筛选以满足用户的使用需求。
- 常规筛选
常规筛选可以显示/隐藏性质。默认的结果列表展示计算的所有性质,此时左边控制栏中为全选的状态。当你不想显示该性质时,取消该性质的选中即可,左边的结果列表将根据控制栏中的选择进行实时显示。在顶部还提供了两个快捷键“全选”和“不选”,方便用户快速选择。
- 高级筛选
在常规筛选的基础上,高级筛选提供了范围筛选,可进一步筛选出某性质指定范围内的分子,以排除不符合预期结果的分子。
(3) 属性解释
把鼠标移入每个性质的名称上,可查看该属性对应的解释。
(4) 排序
点击结果列表中的性质名字可重新排序,如 F(20%),点击一次为升序,再点一次为降序,再点一次即恢复原始排序。
(5) 单个分子详情页
当你想查看某一个分子的所有性质时,可以在结果列表通过点击该分子结构进入分子详情页。
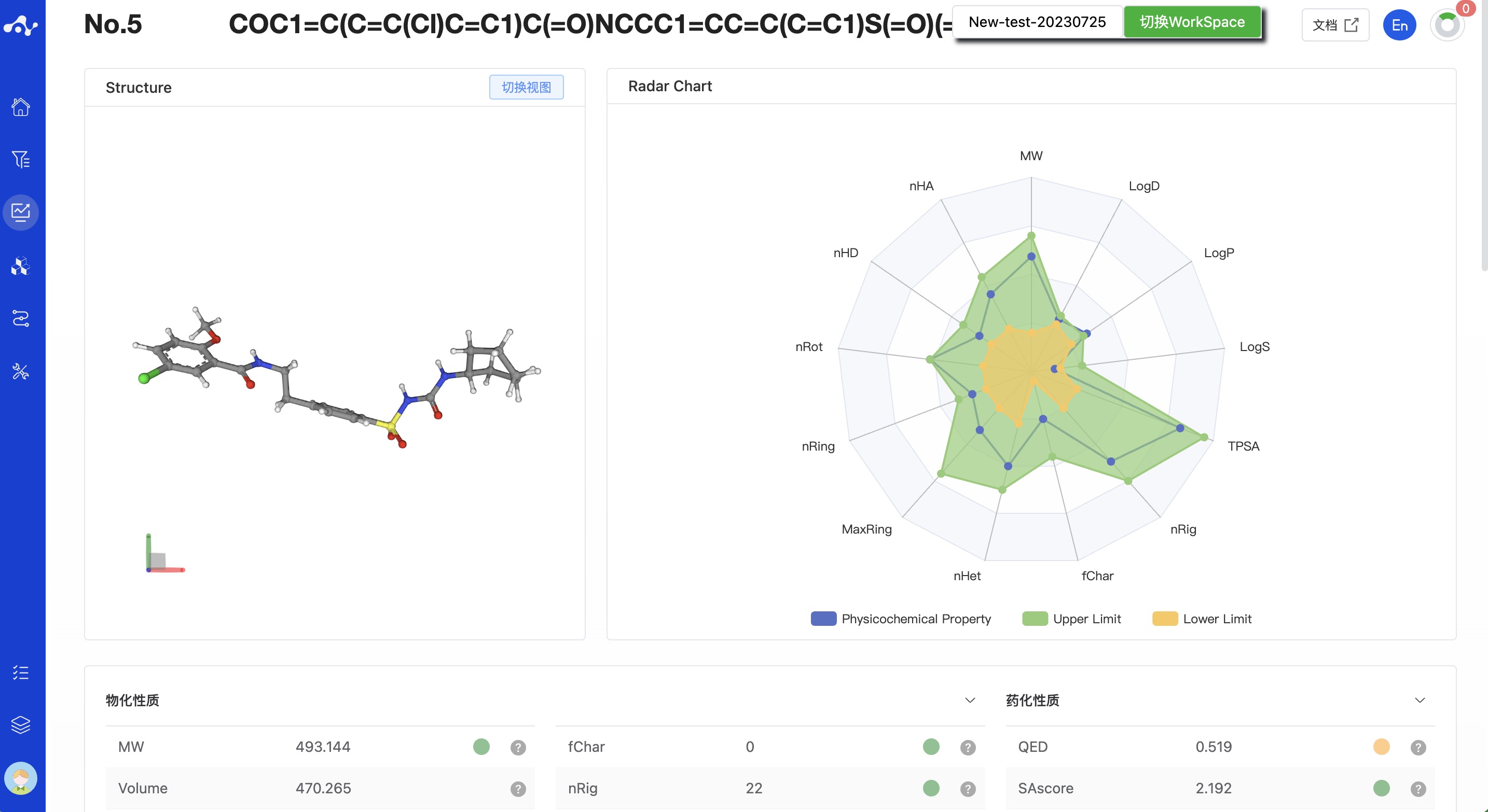
图 8. 分子详情页面-MERT 模型
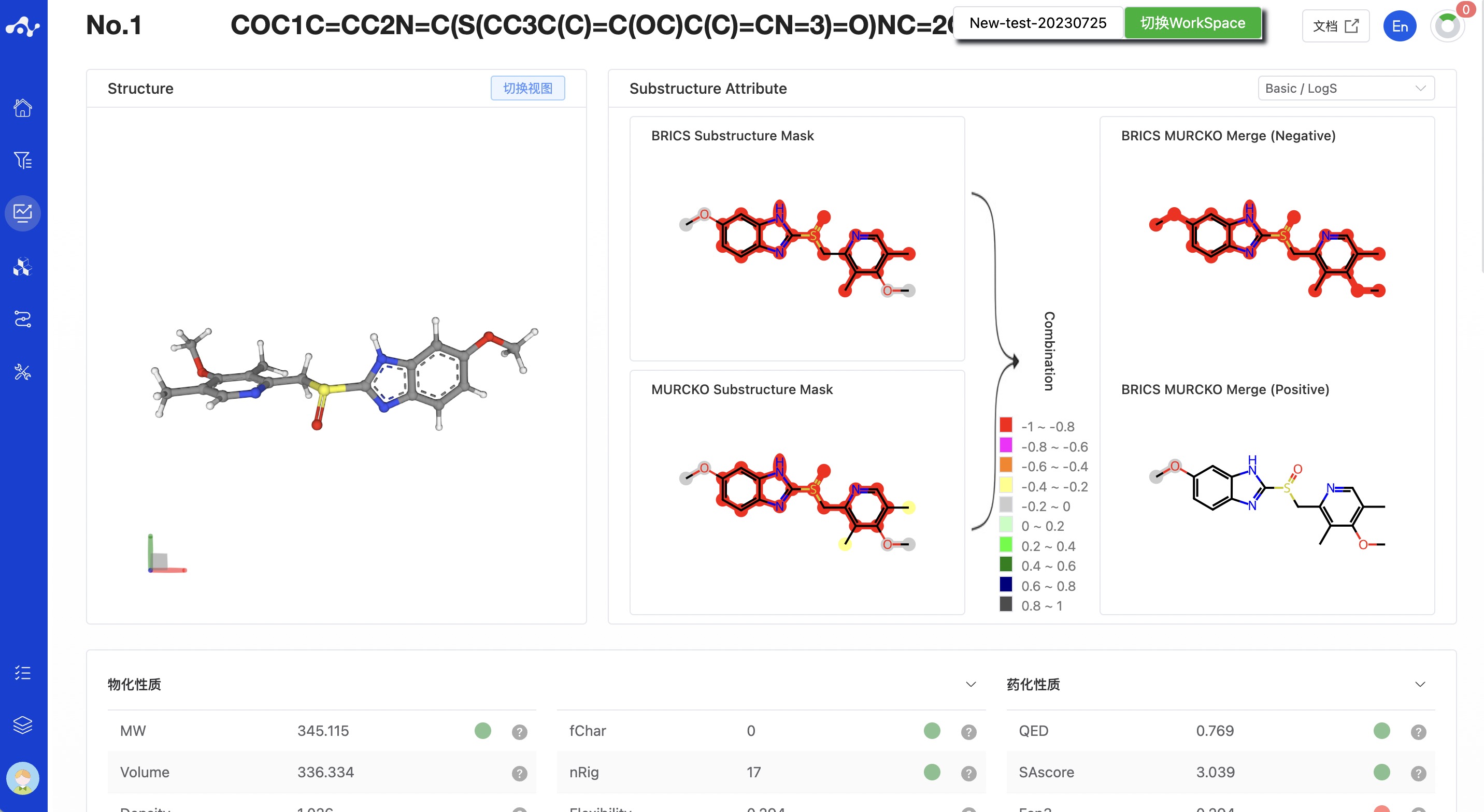
图 9. 分子详情页面-MGA 模型(计算了子结构可解释性)
(6) 保存
点击“保存”,系统将弹出下拉框让你选择保存的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据你的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至数据中心。
(7)下载
点击“下载”,系统将弹出下拉框让你选择下载的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据你的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至你的本地设备上。
(8) 创建新任务
创建新任务的前提是先保存数据为一个文件,在未执行保存操作之前,该按钮为禁用状态,当机遇结果保存了新的文件后,该按钮为可用状态。此时点击该按钮,系统将弹出下拉框让你选择需要计算的工,点击后页面将立即开一个新的标签页,并将你保存的数据集一并带过去,在调整参数后即可提交新的任务。
(9) 隐藏无效分子
针对 SMILES 错误或者后台无法解析的分子,算法无法进行正确的计算,在这种情况下,任务不受影响,但分子被定义为无效分子。用户可以通过“隐藏无效分子”的按钮,快速过滤掉该部分的分子。
(10)卡片页面
在卡片页面,我们提供了一个更加简洁的方式查看结果,你只能看到该分子的结构及 3 个基础的性质:MW、SA 和 QED。
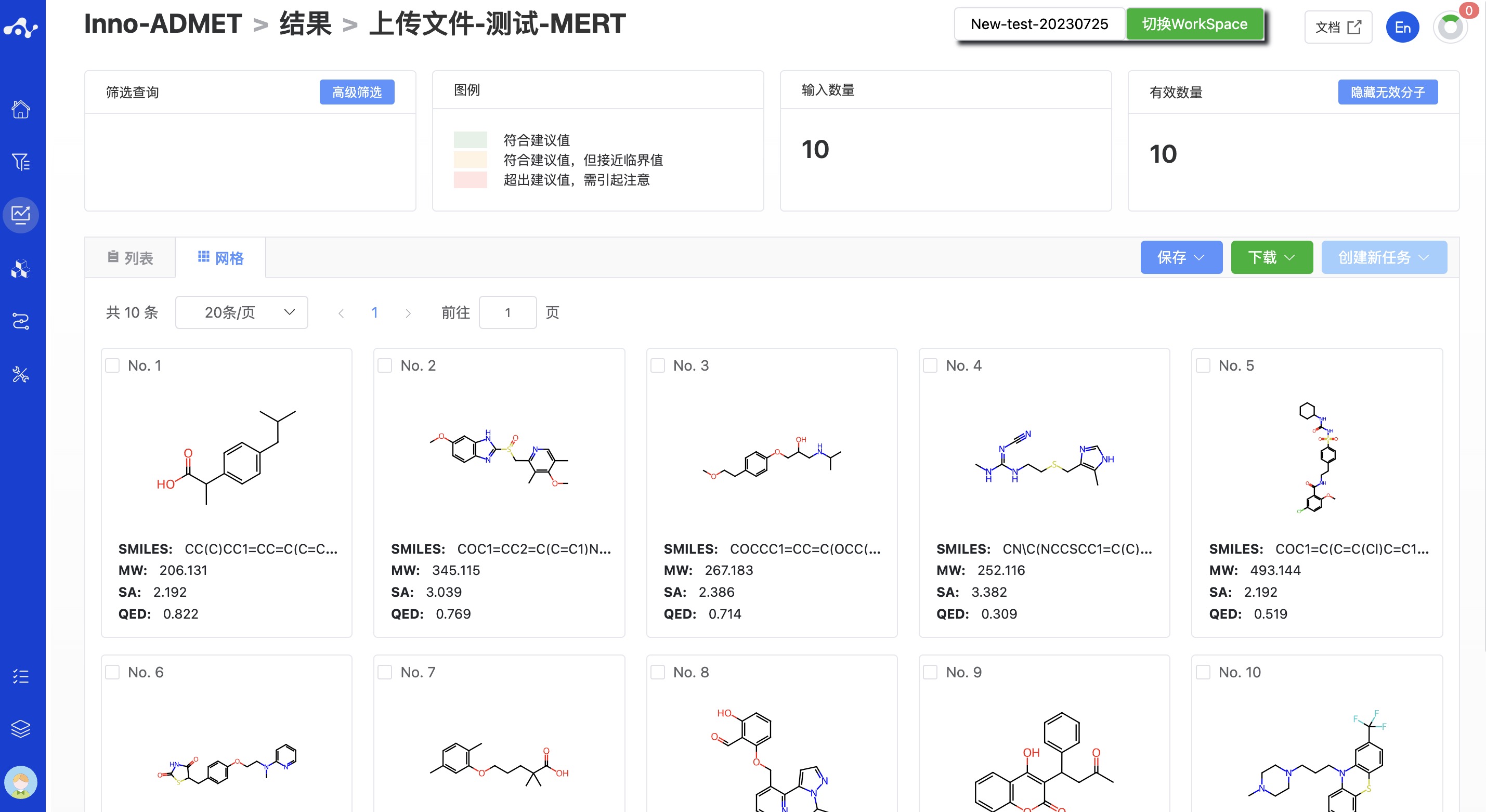
图 10. 结果的网格页面
4. 相关算法介绍
系统提供了两个不同的模型来计算成药性性质,分别为基于预训练的 MERT 模型和基于 GNN 子结构可解释性的 MGA 模型。Inno-ADMET(MERT)和 Inno-ADMET(MGA)建模框架如图 1 和 2 所示。
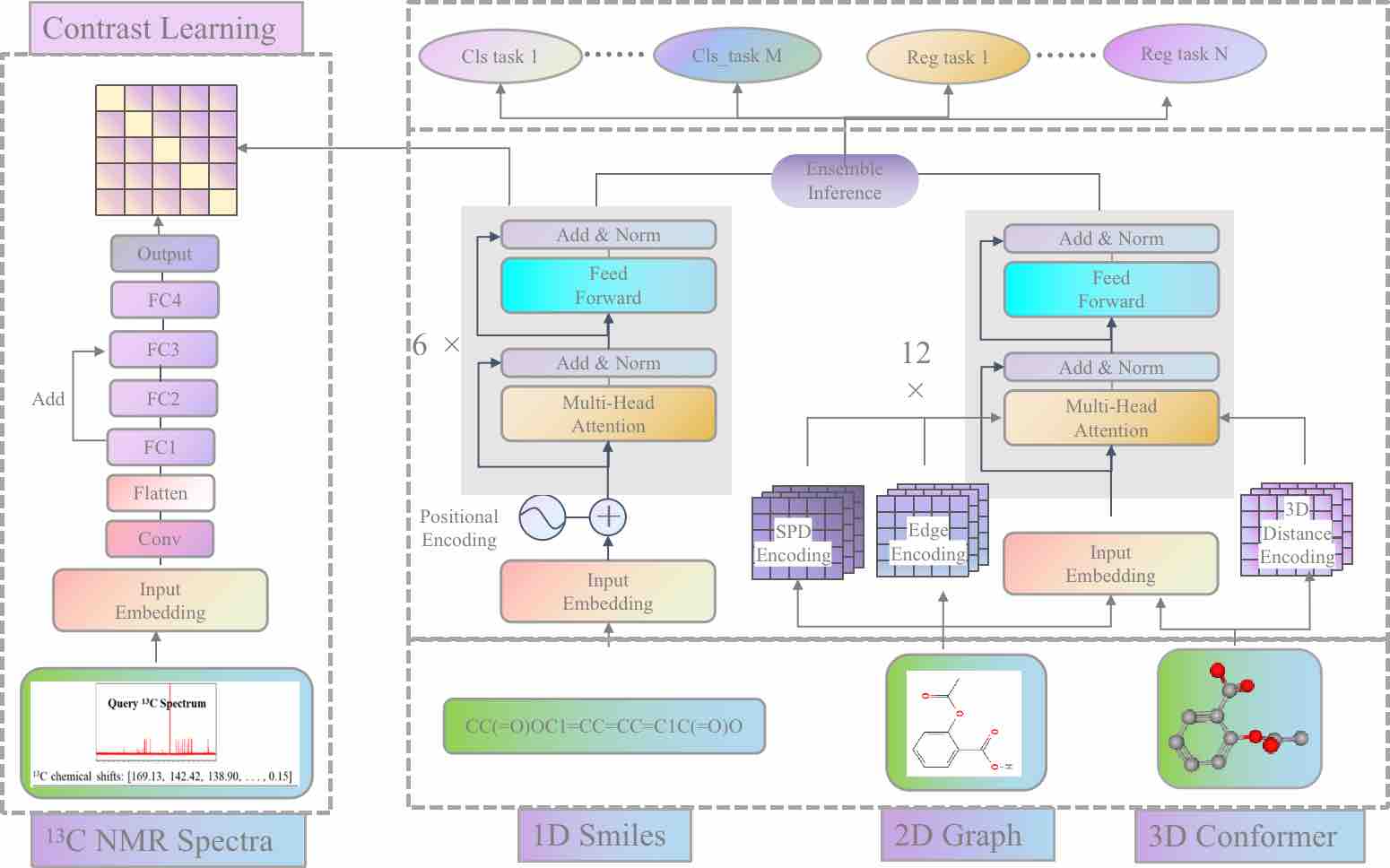
图 11. Inno-ADMET(MERT)建模框架
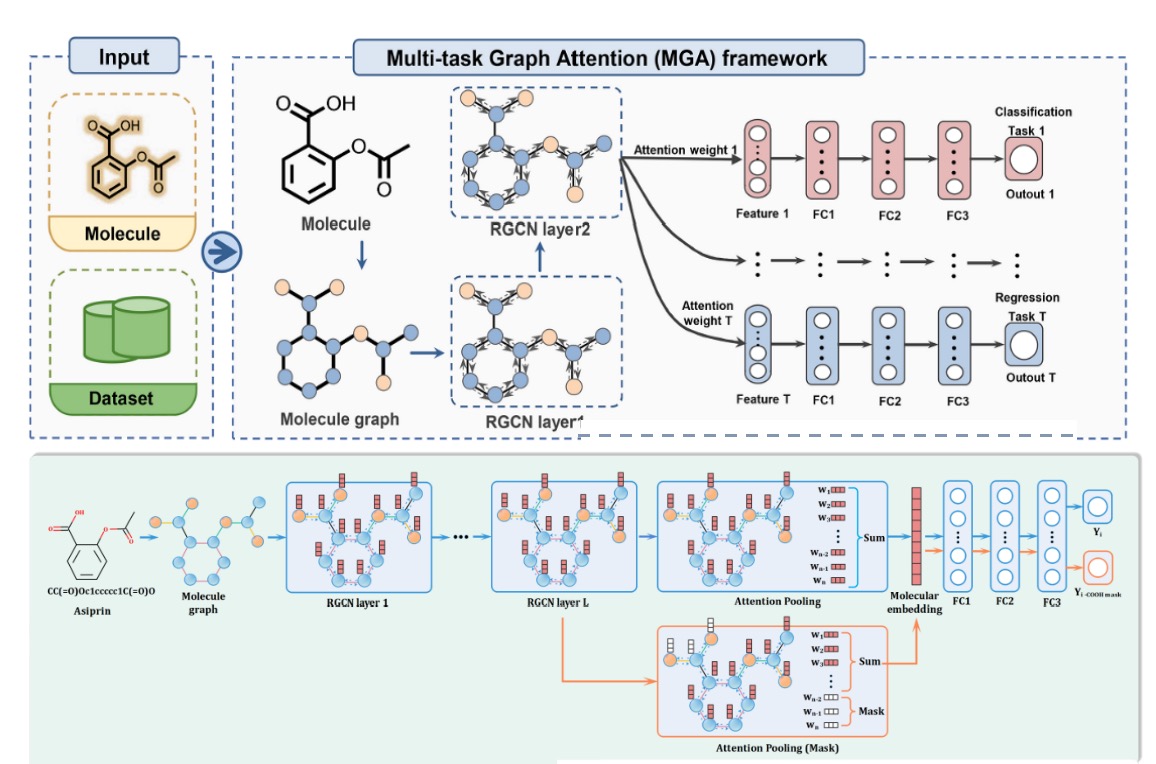
图 12. Inno-ADMET(MGA)建模框架
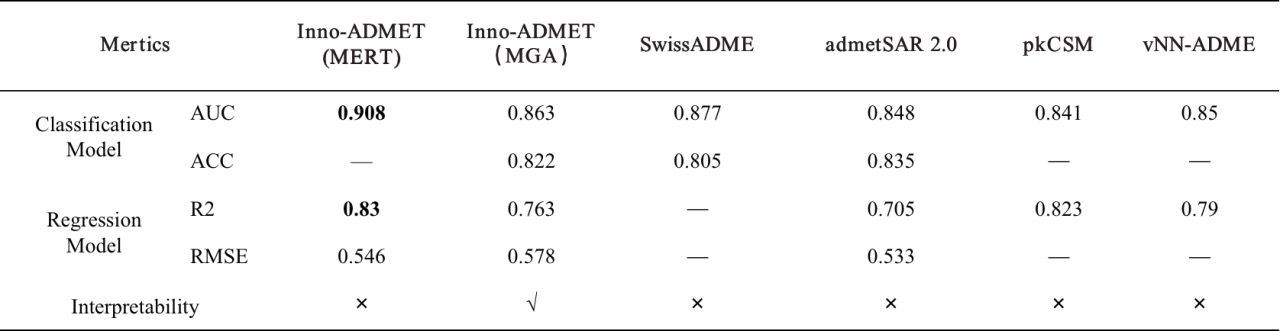
除了我们的 ADMET 模块,同类型计算 ADMET 性质的平台还有 Swiss-ADME、admetSAR 2.0、FAF-Drugs4、pkCSM 和 vNN-ADMET,我们详细比较了这些平台之间的差异,其详细信息汇总在表 1 中。经详细比较得知,这些平台工具各有侧重点,比如 Swiss-ADME 可以提供 logP 和 logS 的多种计算方法以及各种药物相似性规则;admetSAR 2.0 包括最多样化的代谢特性;FAF-Drugs4 首次尝试在 ADMET 评估中加入毒物规则;而 vNN-ADME 中可用的预测性质相对有限。相比之下,ADMET 模块可用于预测分子质量的每个重要方面,涵盖药物化学家感兴趣的主要终点。尤其是平台提供的一些毒性子结构规则、毒性途径和一些药物化学性质预测是独一无二的工具。MERT 模型和 MGA 模型预测结果如表 2 所示。从模型评估指标上来看,Inno-ADMET (MERT) 比其他平台准确率更高;从结果可解释性的角度来看,Inno-ADMET (MGA) 更具优势。 另外从速度上来说,Inno-ADMET (MERT)的速度要优于 Inno-ADMET (MGA),Inno-ADMET (MERT)的速度达到了 2.35ms/分子。
表 1. ADMET 模块与其他基于 web 的工具的主要特性比较。“+”表示支持该特性,每多增加一个“+”表示支持度更高。

表 2. 各个平台的平均评估结果
5. 相关文献
[1] Chemistry-intuitive explanation of graph neural networks for molecular property prediction with substructure masking. Wu Z, Wang J, Du H, et al. Nat Commun. 2023;14(1):2585. doi:10.1038/s41467-023-38192-3
[2] ADMETlab 2.0: an integrated online platform for accurate and comprehensive predictions of ADMET properties. Xiong G, Wu Z, Yi J, et al. Nucleic Acids Res. 2021;49(W1):W5-W14. doi:10.1093/nar/gkab255
[3] ADMETlab: a platform for systematic ADMET evaluation based on a comprehensively collected ADMET database. Dong J, Wang NN, Yao ZJ, et al. J Cheminform. 2018;10(1):29. Published 2018 Jun 26. doi:10.1186/s13321-018-0283-x
