蛋白预处理
1. 蛋白预处理概述
蛋白质预处理是计算机辅助药物设计中的一个重要环节,它涉及对蛋白质模型的准备和优化,以便进行后续的分析和模拟。为了方便用户更好的使用 DrugFlow 中与蛋白相关的功能,为此我们提供了一个交互式编辑蛋白结构及其预处理的功能。蛋白预处理功能包括:1. 去除蛋白质结构中的无关成分(例如水分子、离子等)以及可能对后续分析造成干扰的成分;2. 补全缺失的原子或残基;3. 错误残基结构的修正;4. 调整质子化状态并优化氢网络;5. 对蛋白质进行最小能量优化等。
总的来说,蛋白质预处理为后续的药物设计步骤(如对接、分子动力学模拟等)提供了准确、可靠的蛋白质模型,有助于提高药物设计的质量和准确。
2. 使用说明
蛋白预处理的计算页面分为左右两个区域,左边为蛋白可视化区域,右边为设置计算参数,整个蛋白预处理分为四个步骤:保留结构-修复错误结构-添加缺失残基-优化,每个步骤中都有一些参数供你选择。
图 1. 蛋白预处理计算空白页面
(1) 保留结构
此步主要输入蛋白文件并且选择需要保留的成分。这里的成分包括三个部分:蛋白链,小分子(包括小肽、小分子和离子),水分子。
- 输入蛋白
输入蛋白文件的方式有两个,分别为上传文件或从数据中心选择。
- 上传文件
复选框选中“上传文件”,通过点击下方按钮选择本地文件即可。选择完文件以后,该文件名称将显示在按钮上,右边将显示文件内容。 关于上传的文件只支持.pdb 格式。
- 数据中心
复选框选中“数据中心”,通过点击下方按钮页面出现弹窗,点击文件名称来选择数据中心的数据,点击完之后弹窗消失,即可提交任务。
- 选择需要保留的蛋白链
此处默认显示上传的.pdb 文件中所有链的信息,默认所有链都被勾选。当某条链被取消勾选后,蛋白可视化区域的内容与将看不到该链。
- 选择需要保留的小分子和水分子
此处默认展示 pdb 文件中所有的小分子及其 5Å 范围内的水分子。系统会默认删除 5Å 范围外的水,而 5Å 范围内的水由用户自行决定是否删除。其中具有水桥作用的水分子做了特殊标记,你可以通过【快速删除】按钮一步删除没有水桥作用的水分子。
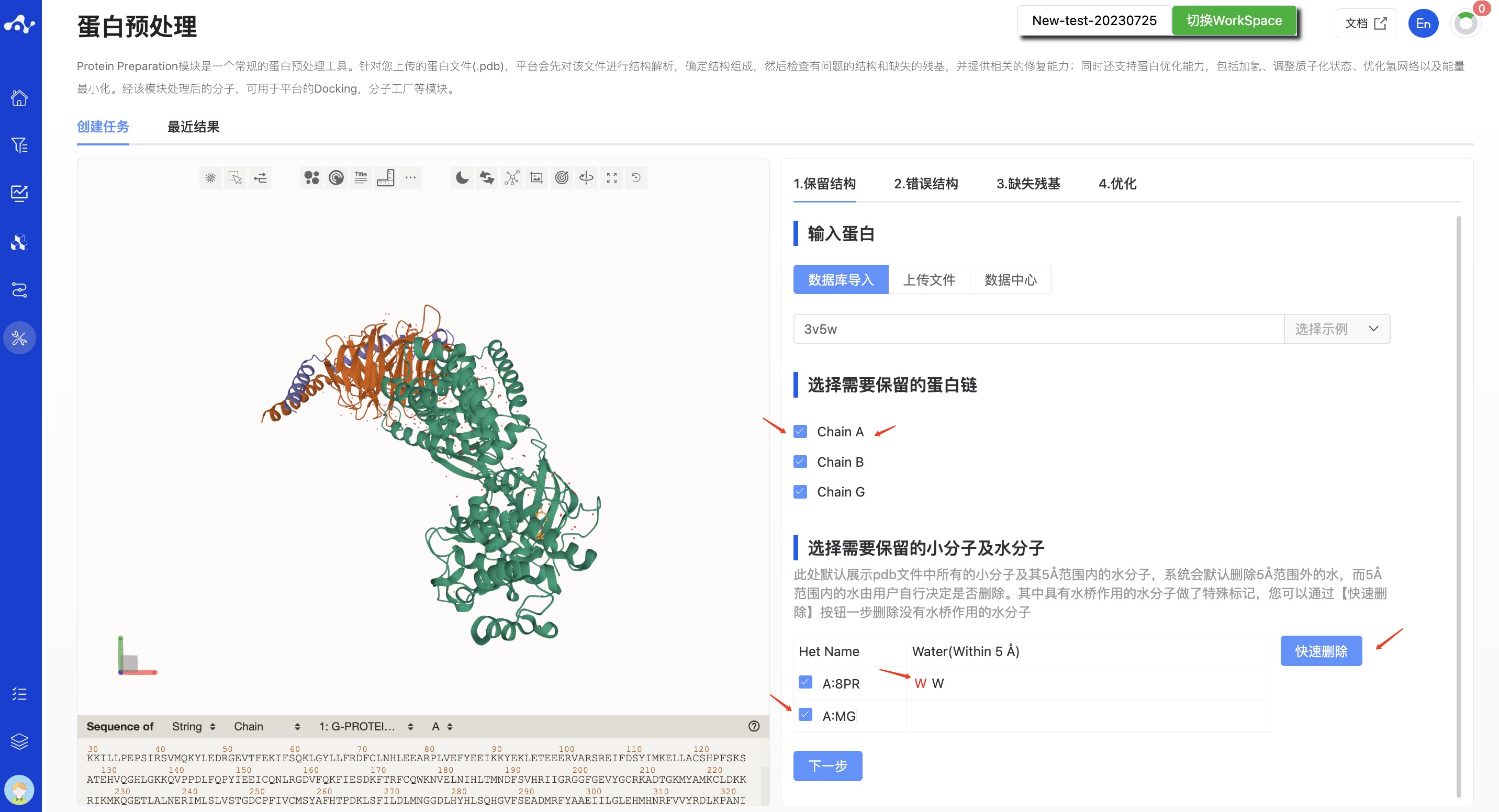
图 2.蛋白预处理-保留结构计算页面
(2) 错误结构
此步骤主要用于检查和修复错误的残基信息。每个错误的残基都给出了具体的错误类型以及错误详情。该内容以表格的形式展示,每一行都是可点击的状态,点击该行,则左侧可视化区域则对其进行快速聚焦。
系统默认勾选所有有问题的项目,你也可以取消勾选,在该状态下进行结构修正时,未勾选的错误结构将不会被修正。勾选的项目中,能够被纠正的项目则不再显示在表格中,而不能被修复的项,则仍旧保留在页面上,用户可以手动修改错误。
点击【纠正】按钮,按钮上会显示“运行中”的交互,直到运行结束,该交互消失,右边蛋白可视化区域将显示修正后的蛋白。只有点击【纠正】以后,才能点击【下一步】。
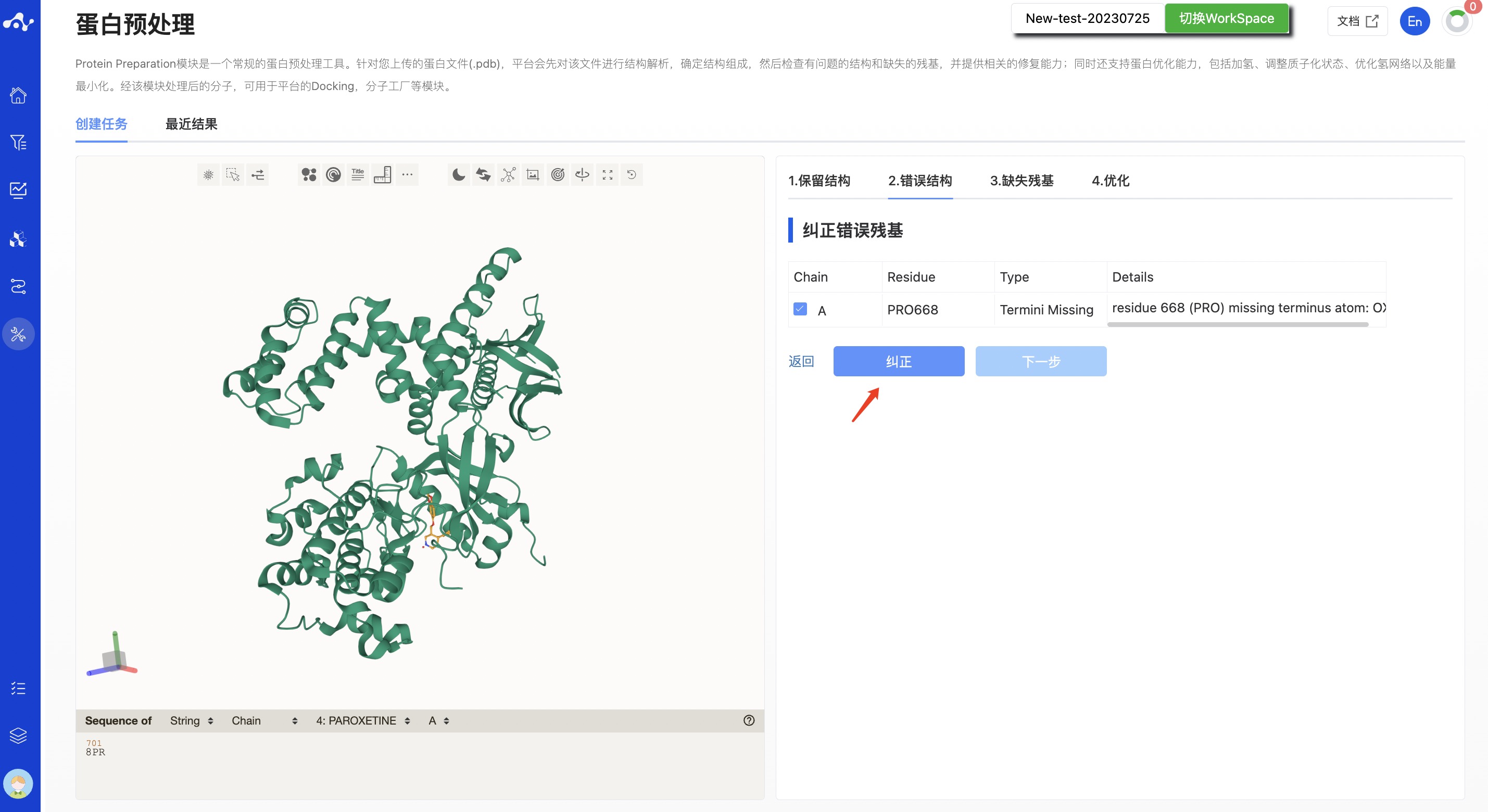
图 3.蛋白预处理-错误结构计算页面
(3) 缺失残基
此步骤用于检查和修复 pdb 文件序列中缺失的残基序列。该内容以表格的形式展示,每一行都是可点击的状态,点击该行,则左侧可视化区域则对其进行快速聚焦。
系统默认勾选所有有缺失的片段,你也可以取消勾选,在该状态下进行结构修正时,未勾选的残基序列将不会被修正。勾选的项目中,能够被纠正的项目则不再显示在表格中,而不能被修复的项,则仍旧保留在页面上,用户可以手动修改错误。
点击【纠正】按钮,按钮上会显示“运行中”的交互,直到运行结束,该交互消失,右边蛋白可视化区域将显示修正后的蛋白。
你也可以不修复缺失残基,直接点击【下一步】。
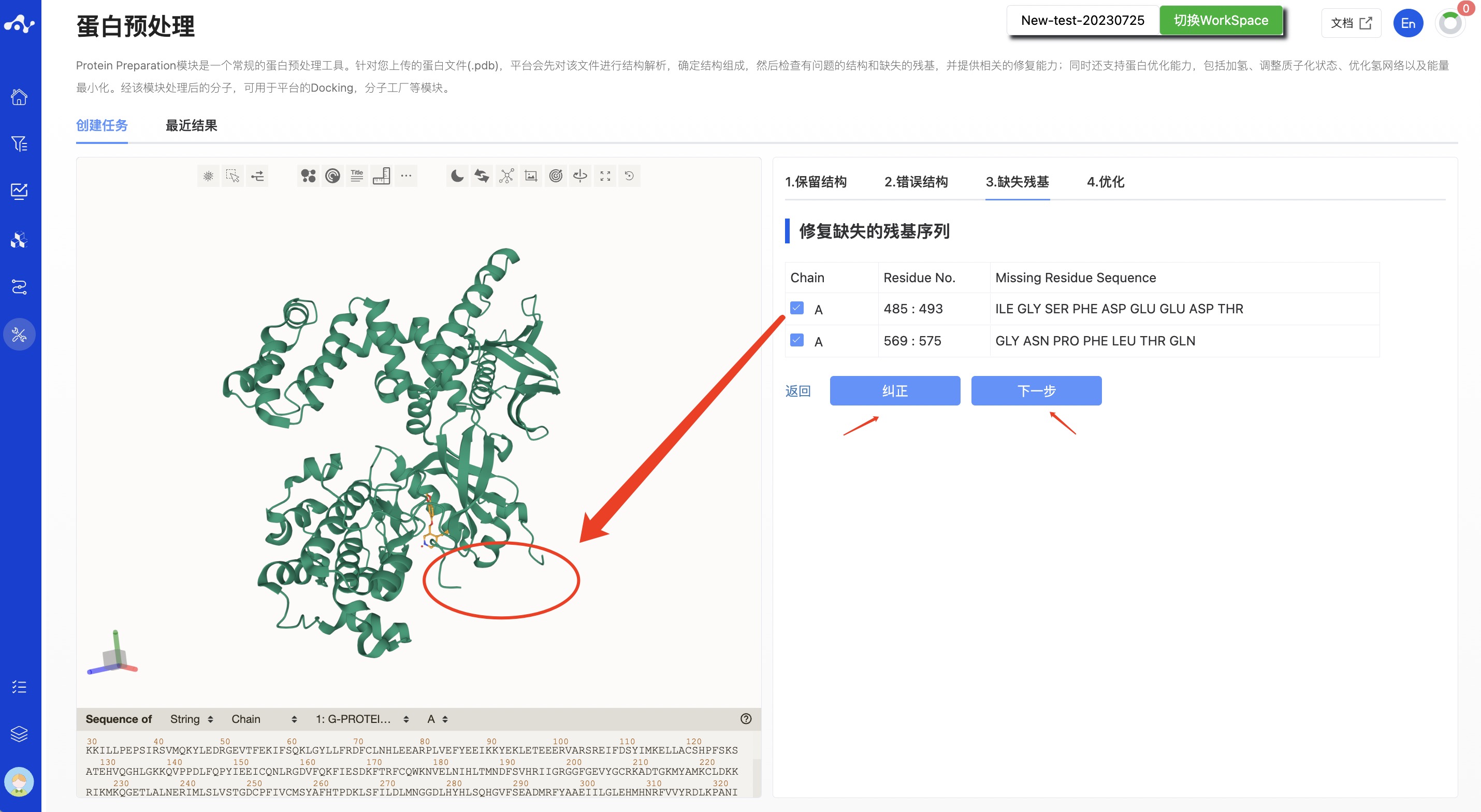
图 4.蛋白预处理缺失残基计算页面
(4) 优化
- 二硫键
系统提供二硫键的功能。此处将显示蛋白中可能的所有二硫键的 pair,默认选中 1.5~3Å 范围的二硫键,基于您的研究目的可自行调整,选中的二硫键 pair 将会在蛋白预处理时保留,此操作对蛋白构象影响较大,请谨慎选择。如果不需要该功能,也可以直接关闭。
- 蛋白优化
蛋白加氢:必选项;
调整质子化状态:可选项,默认为勾选状态,且 pH 为 7.4;
氢网络的优化:可选项,默认为勾选状态;
能量最小化:可选项,默认为勾选状态,且选择的为 AMBER ff14SB 力场;
AMBER ff14SB(推荐使用). ff14SB 是 AMBER 程序包中的一个蛋白质力场参数集,用于描述生物分子中的原子相互作用。它是 AMBER14 程序包中的一种特别适用于蛋白质体系的力场参数集,包括描述蛋白质中氨基酸侧链和蛋白质折叠中重要残基之间相互作用的额外参数。AMBER ff14SB 在描述蛋白质的构象和动力学性质方面具有较高的精度和可靠性。
AMBER ff15IPQ. ff15ipq 是 AMBER14 程序包中的一种改进的蛋白质力场参数集,相比于 AMBER ff14ipq 具有更高的精度和可靠性。AMBER ff15ipq 包括更多的偏极化效应和氢键参数,可以更准确地描述蛋白质的电子结构。
AMBER96. AMBER96 是 AMBER 程序包中的早期版本,经过多年的发展和优化,现在已有更新的版本,如 AMBER14 和 AMBER18 等。但是,AMBER96 仍然被广泛应用于生物分子模拟领域,特别是对于早期的研究和一些经典案例的模拟。
AMBER99SB. AMBER99sb 是 AMBER99 力场的一个改进版本,包括了描述蛋白质中氨基酸侧链和蛋白质折叠中重要残基之间相互作用的额外参数。AMBER 在描述蛋白质的构象和动力学性质方面具有较高的精度和可靠性。
CHARMM36. CHARMM36 在描述蛋白质的构象和动力学性质方面具有较高的精度和可靠性。它在蛋白质和蛋白质-配体相互作用等领域的研究中被广泛应用,是生物分子模拟领域中常用的力场参数集之一。
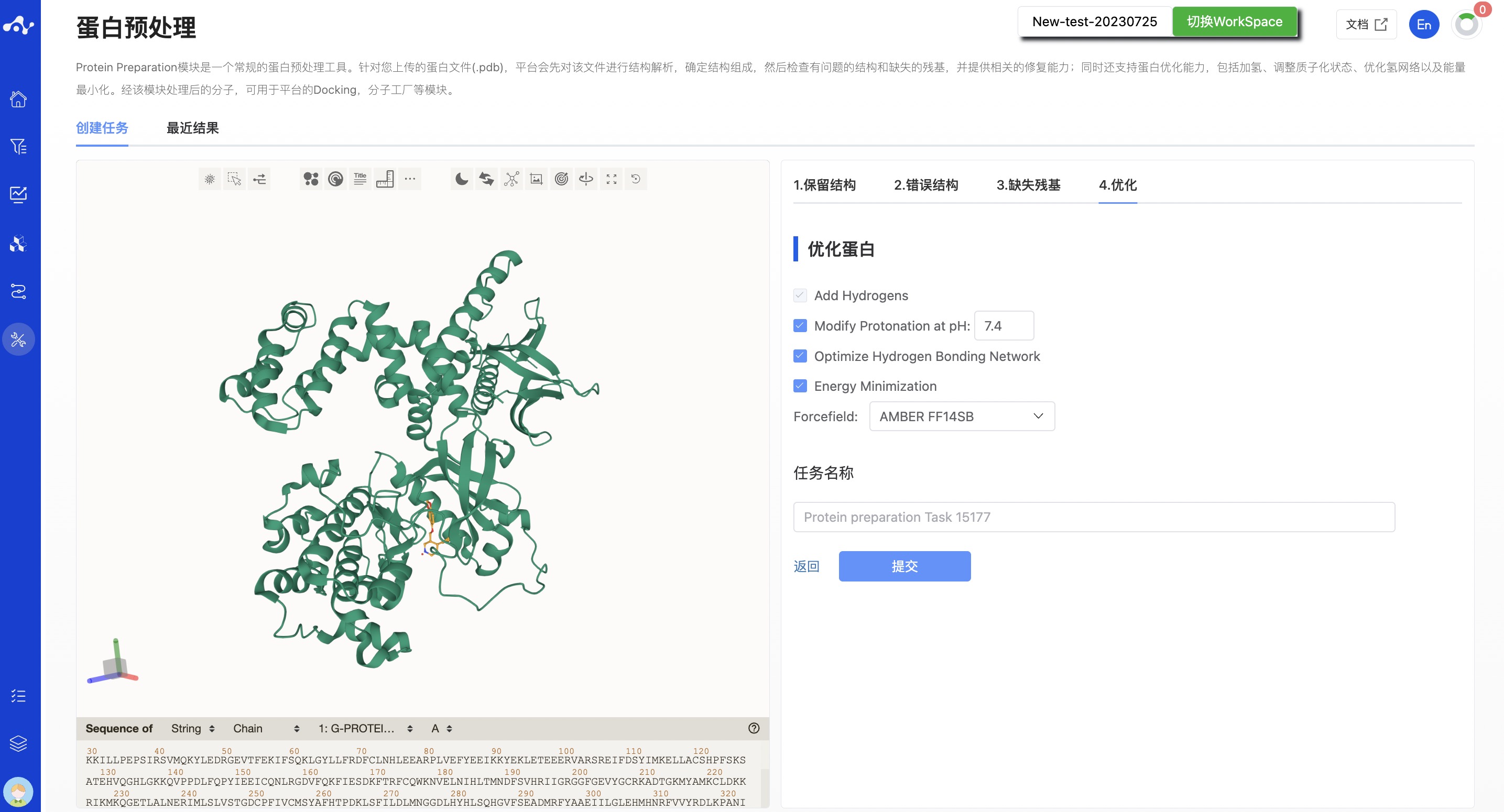
图 5.蛋白预处理-优化计算页面-无二硫键
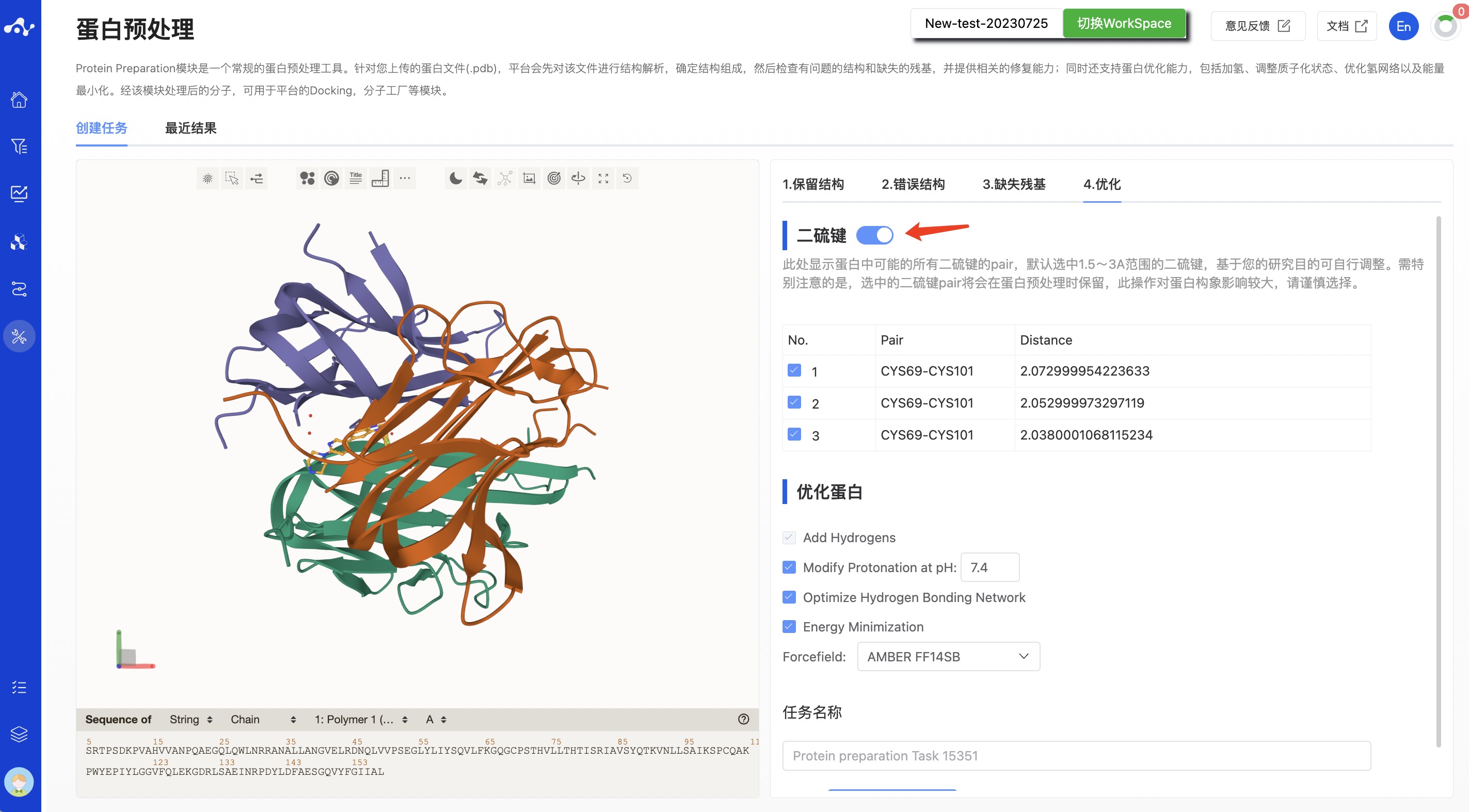
图 6.蛋白预处理-优化计算页面-有二硫键
(5) 运行进度和结果查看
提交任务后,页面会自动跳入当前页面的“最近结果”子页面中,你可以在该页面查看当前模块的任务运行状态(进度条),也可在右上角的“运行中“下拉框中查看所有模块正在运行的任务。
图 7. 查看运行状态和结果
3. 结果分析
蛋白预处理的结果页面主要展示的是预处理后的蛋白。除此之外,我们还提供了保存、下载、创建新任务的功能。
(1) 保存
点击“保存”,系统将弹出下拉框让你选择保存的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据你的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至数据中心。
(2)下载
点击“下载”,系统将弹出下拉框让你选择下载的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据你的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至你的本地设备上。
(3) 创建新任务
创建新任务的前提是先保存数据为一个文件,在未执行保存操作之前,该按钮为禁用状态,当机遇结果保存了新的文件后,该按钮为可用状态。此时点击该按钮,系统将弹出下拉框让你选择需要计算的工,点击后页面将立即开一个新的标签页,并将你保存的数据集一并带过去,在调整参数后即可提交新的任务。
