AI 解谱(NMR)
1. AI 解谱(NMR)概述
基于核磁共振(NMR)方法求解未知化合物的结构是化学科学中一个长期存在的逆问题。发现新的解决结构解析逆问题的方法将有助于研究人员加速化学发现,尤其对于有机化学及天然产物化学的研究具有重要意义。AI 解谱模块主要在于应用 AI 技术来快速解析碳谱,帮助专家识别分子结构。 在此,我们提供了两种不同的方式 CRESS 和 CMGNet 来基于碳谱解析未知化合物的结构。
2. 使用说明
平台提供了两种解谱的方法:CRESS 和 CMGNet。根据选择方法完成相应的输入后,在“任务名称”的文本框里面给任务命名,点击“提交”按钮后进入后台计算步骤。
(1) CRESS
复选框选中“CRESS”,在“输入碳谱”的文本框中输入碳谱报告中的各个化学位移值。
注意:需去除溶剂峰。
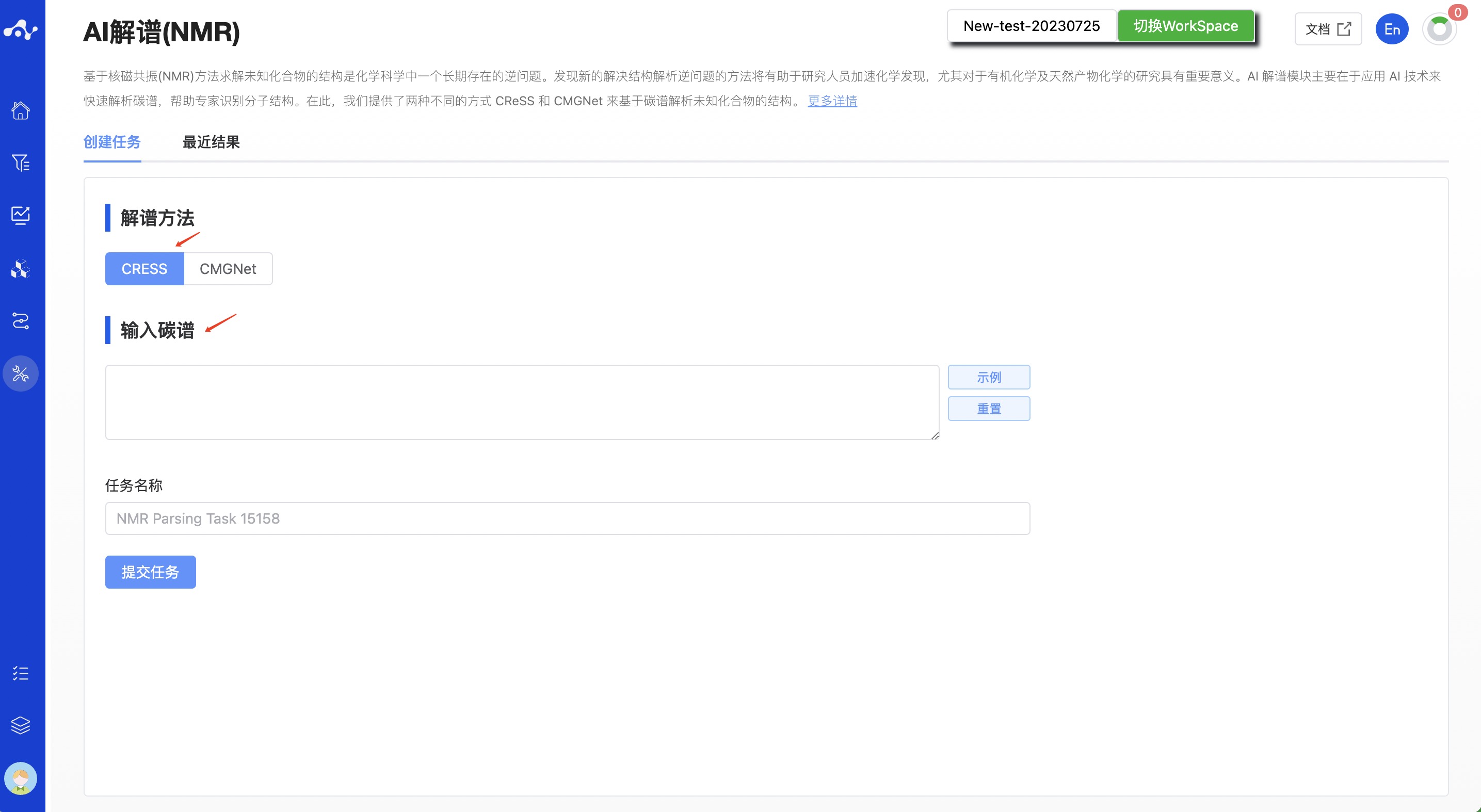
图 1. CRESS 计算页面
(2) CMGNet
复选框选中“CMGNet”,在“请输入碳谱”的文本框中输入碳谱中的各个化学位移值(去除溶剂峰)。CMGNet 与 CRESS 不同,它可以选择性的输入其他两项:
分子式。如果你已通过质谱或其他分析手段得知了该分子的分子式,可在此输入。
已确定的分子片段的 SMILES。➀ 如果你已经知道了未知化合物的部分子结构,可将其 SMILES 输入到文本框,多个 SMILES 换行输入即可。➁ 如果仅知道片段结构不知道 SMILES,可从下面的分子编辑区画分子片段,画完一个点击 SMILES 后面的 SEND 即可将 SMILES 输入至文本框中,清空编辑器后再画第二个分子,以此类推。
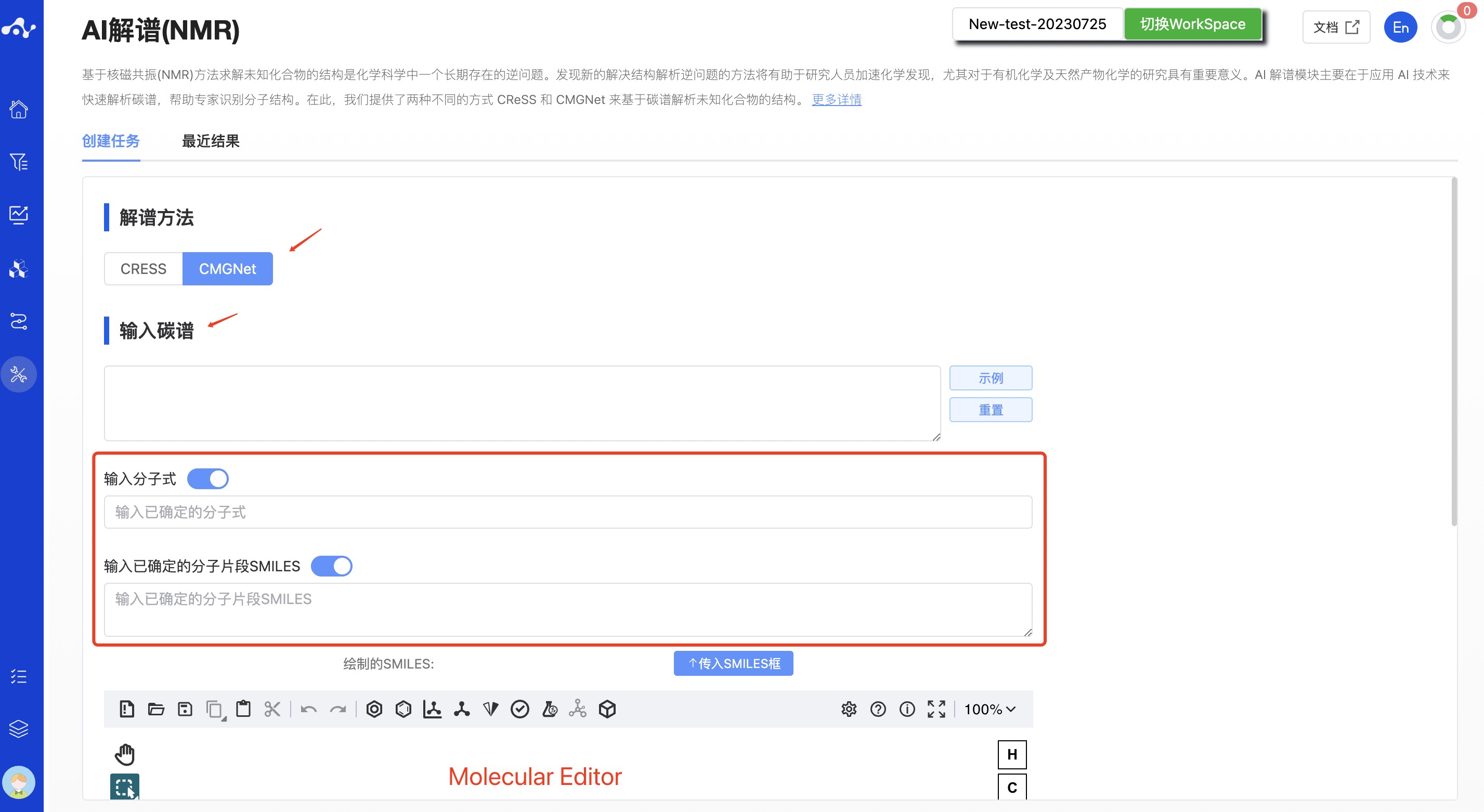
图 2. CMGNet 计算页面
(3) 运行进度和结果查看
提交任务后,页面会自动跳入当前页面的“查看结果”子页面中,你可以在该页面的“结果列表”中查看当前模块的任务运行状态(进度条),也可在右上角的“运行中“下拉框中查看所有模块正在运行的任务。任务完成计算后,页面右上角会弹出卡片以提示你有新的任务已完成,你可以点击卡片中的“查看结果”按钮查看结果,也可以在当前“查看结果”页面上点击“结果详情”按钮查看结果。
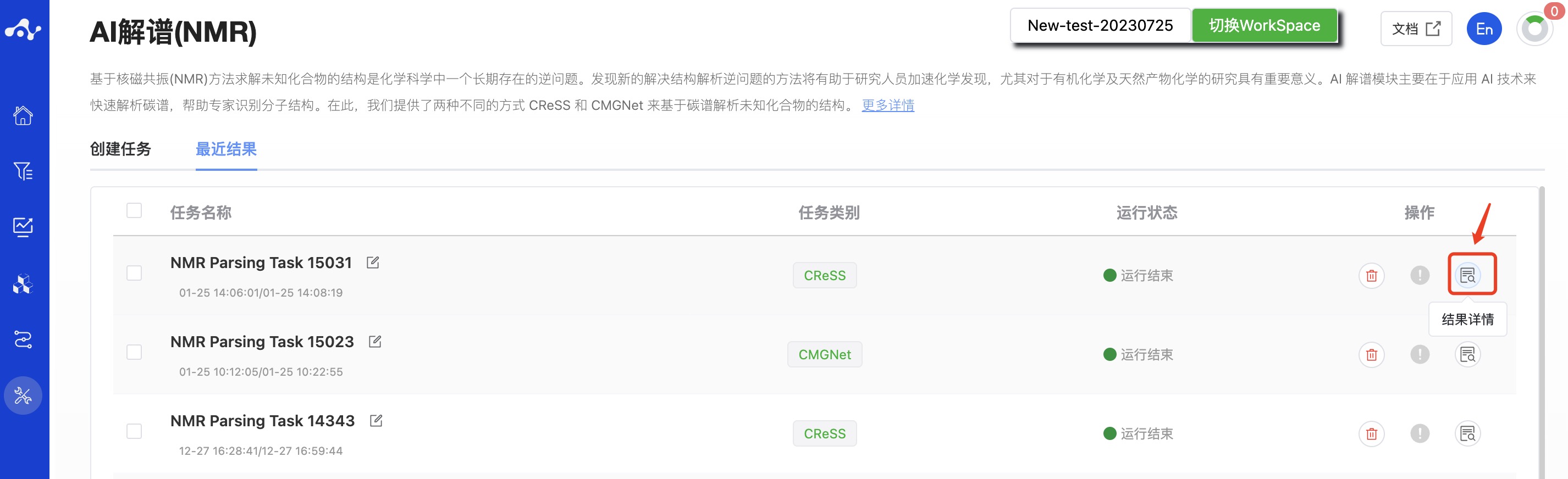
图 3. 查看任务运行状态
3. 结果分析
结果页面由顶部的 Summary 和中部的结果详情区组成。默认状态下结果详情区展示的是结果列表(还可以切换卡片),此时你可以在页面上看到预测的分子结构以及几个简单的性质,并对这些性质进行排序、筛选;同时为了方便用户直观的分析数据,我们给预测结果分配了一个颜色。CReSS 和 CMGNet 的结果页面稍有不同。
CReSS: 当你选择使用 CRESS 计算时,系统会利用 NMR Encoder 将你输入的 13C NMR 化学位移数据转换成 NMR 特征向量,并和分子结构库中预先存储的分子的 SMILES 特征向量之间进行余弦相似度计算,因此最终检索出的每个候选结构都会附带一个余弦相似度数值(cosine Similarity score),结果也是默认根据该值进行排序的,Similarity score 越大,代表该候选结构是正确结构的可能性也就越大。
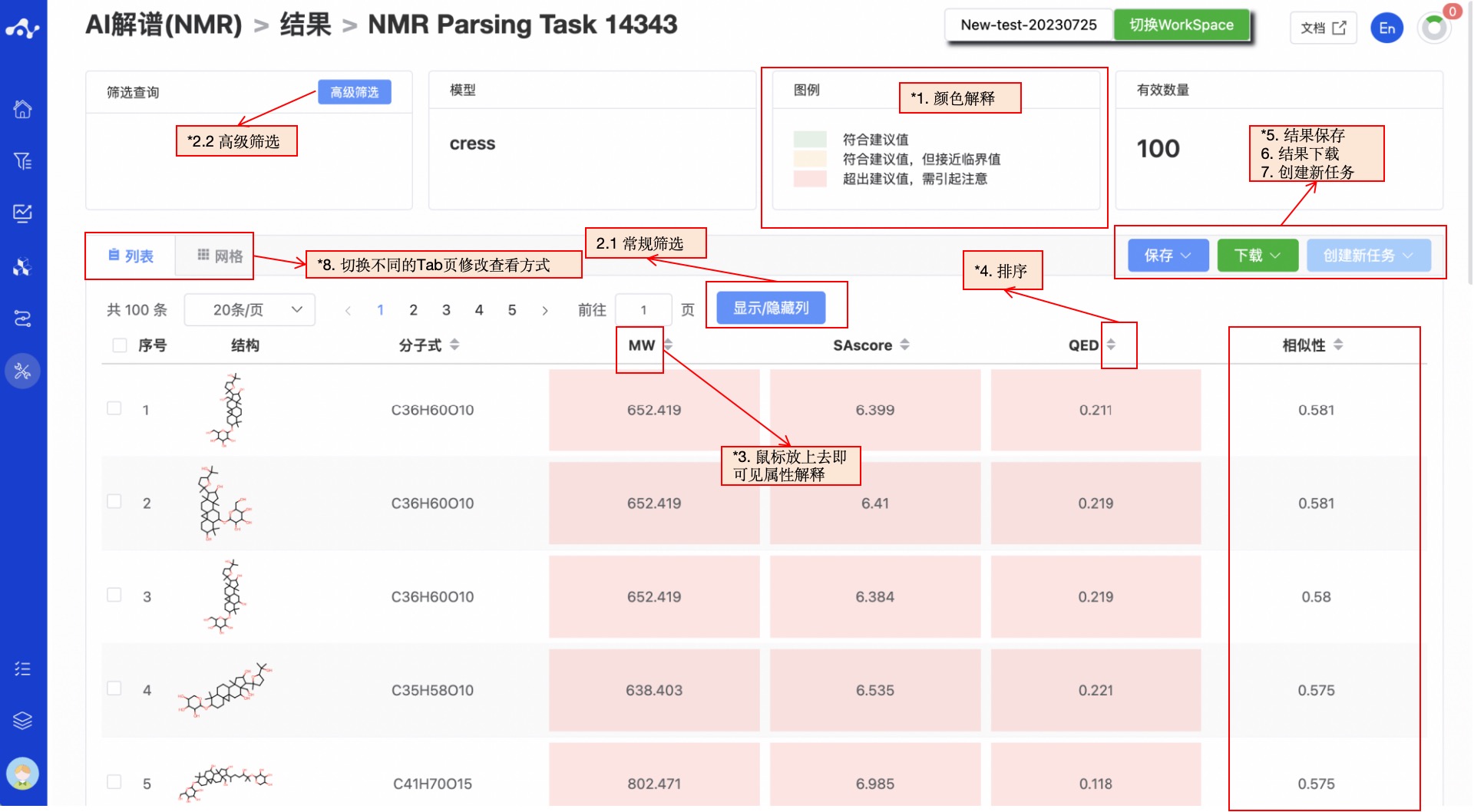
图 4. CRESS 结果页面
CMGNet: CMGNet 则通过候选结构的排名体现该结构是正确结构的可能性的相对大小,即排序越靠前的分子表明其可信度越高。
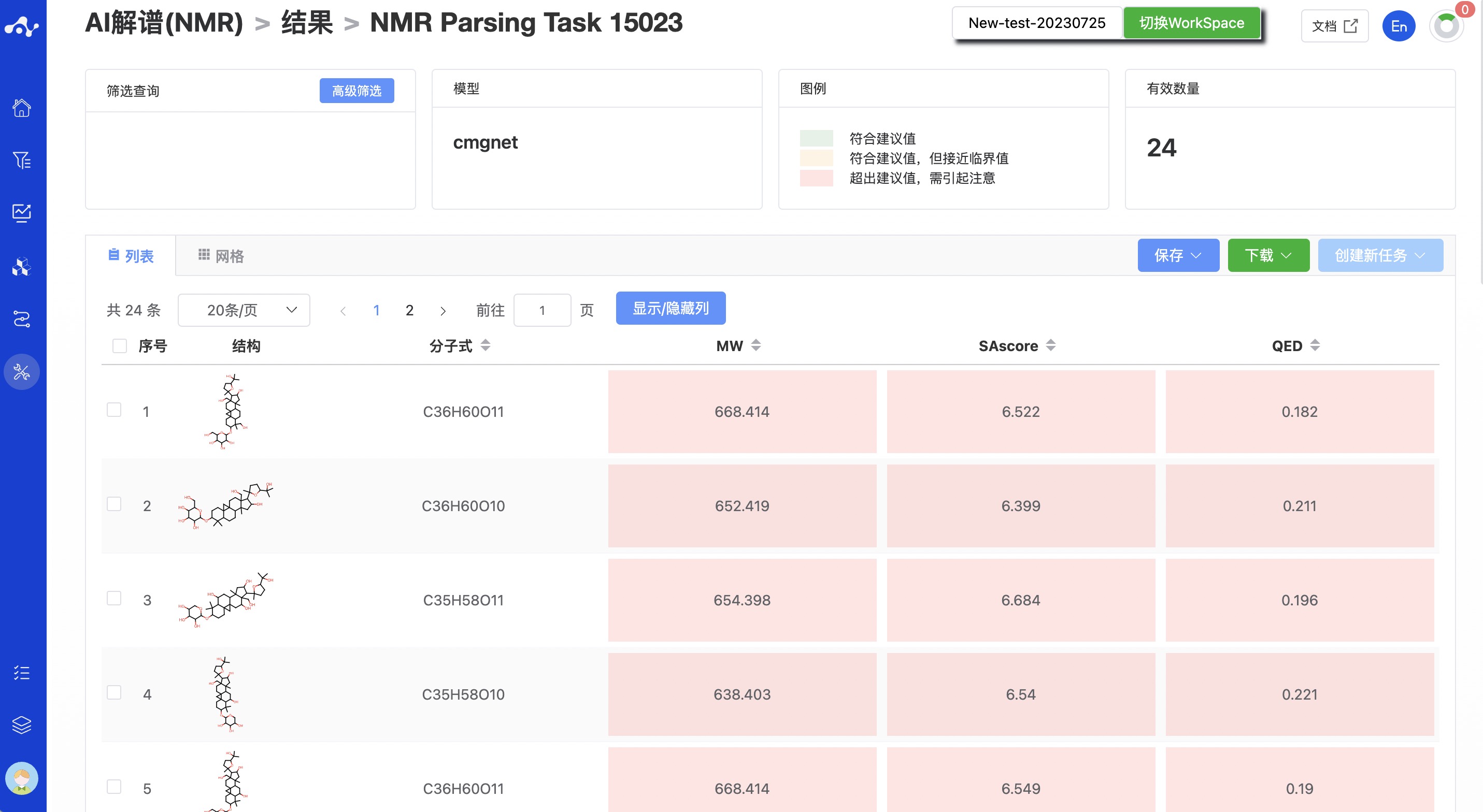
图 5. CMGNet 结果页面
(1) 颜色定义
由于不同的性质有不同的建议取值区间,因此我们通过颜色对预测结果做了直观的评估。颜色设置的规则如下:
绿色:符合建议值
橙色:符合建议值,但接近临界值
红色:超出建议值,需引起注意
通过以上颜色区分,你可以更直观的了解了解分子的整体评价结果。
(2) 筛选
平台提供了常规筛选和高级筛选以满足用户的使用需求。
- 常规筛选
点击列表中的“显示/隐藏性质”按钮即可实现常规筛选。默认的结果列表展示计算的部分性质。当你想要显示/隐藏某些属性时,你可以点击显示/隐藏按钮,页面会从页面的右侧样式弹出一个抽屉,你可以勾选/取消勾选某些属性,左边的结果列表会根据选择实时显示出来。顶部还提供了“全选”、“不选”和“默认”三个快捷键,方便用户快速选择。
- 高级筛选
在常规筛选的基础上,高级筛选提供了范围筛选,可进一步筛选出某性质指定范围内的分子,以排除不符合预期结果的分子。
(3) 属性解释
把鼠标移入每个性质的名称上,可查看该属性对应的解释。
(4) 排序
点击结果列表中的性质名字可重新排序,如 Log S,点击一次为升序,再点一次为降序,再点一次即恢复原始排序。
(5) 保存
点击“保存”,系统将弹出下拉框让你选择保存的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据你的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至数据中心。
(6)下载
点击“下载”,系统将弹出下拉框让你选择下载的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据你的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至你的本地设备上。
(7) 创建新任务
创建新任务的前提是先保存数据为一个文件,在未执行保存操作之前,该按钮为禁用状态,当机遇结果保存了新的文件后,该按钮为可用状态。此时点击该按钮,系统将弹出下拉框让你选择需要计算的工,点击后页面将立即开一个新的标签页,并将你保存的数据集一并带过去,在调整参数后即可提交新的任务。
(8) 卡片页面
在卡片页面,我们提供了一个更加简洁的方式查看结果,你能看到该分子的结构及几个基础的性质:MW、log P、SA 和 QED。
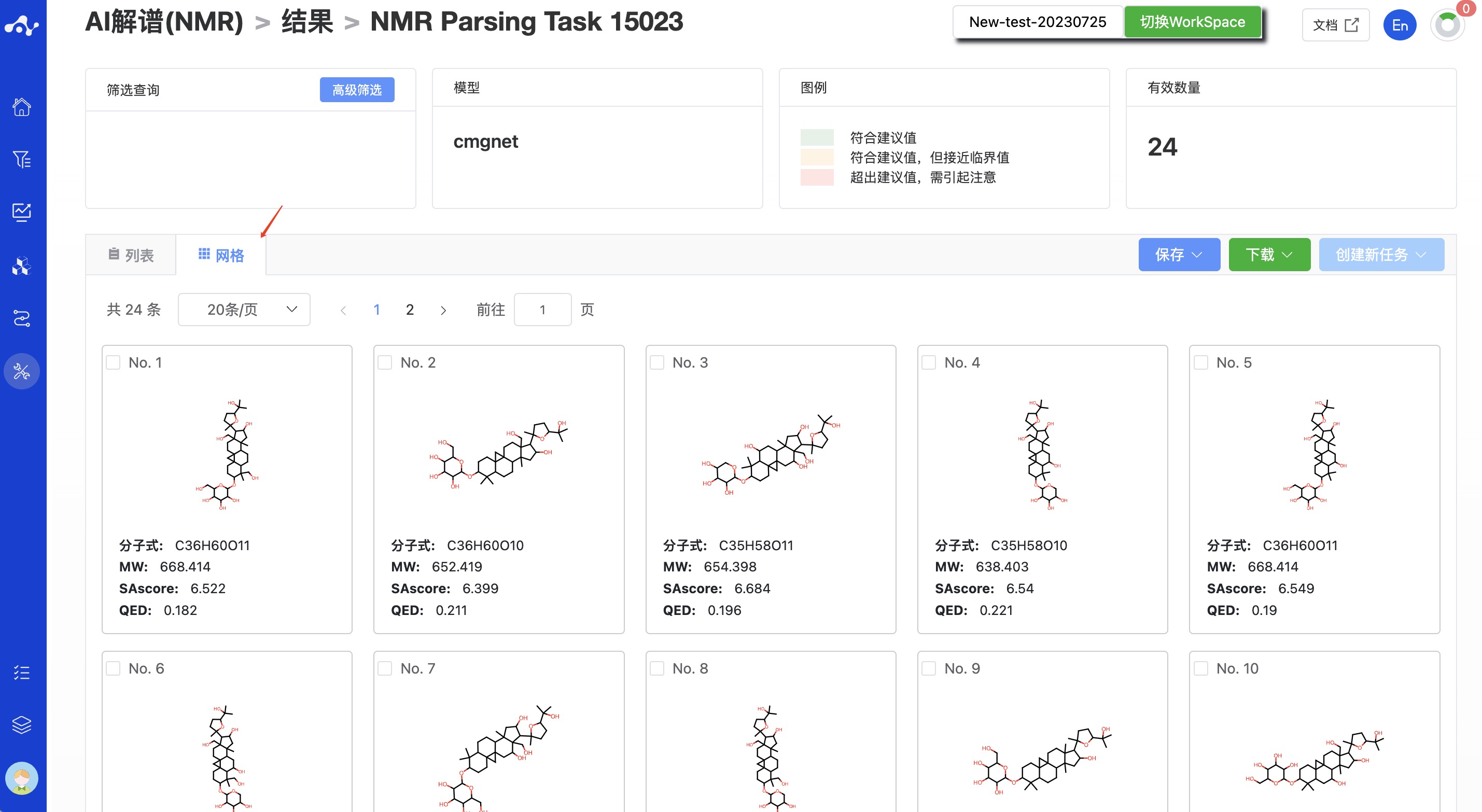
图 6. 结果的网格页面
4. 相关文献
(1) CReSS
CReSS(Cross-modal Retrieval between Spectrum and Structure)是一个基于深度对比学习的跨模态碳谱-结构检索方法。我们采用对比学习训练了 NMR 编码器和 SMILES 编码器来挖掘核磁碳谱与分子结构的内在关系并比较他们的相似性(图 7),并构建了包含上千万化合物及其结构特征的分子结构库,实现了直接在分子结构库识别未知化合物的核磁碳谱。经测试,CReSS 在识别化合物结构任务上展现出了卓越的性能,且能够辅助结构解析和结构纠错(图 8)。
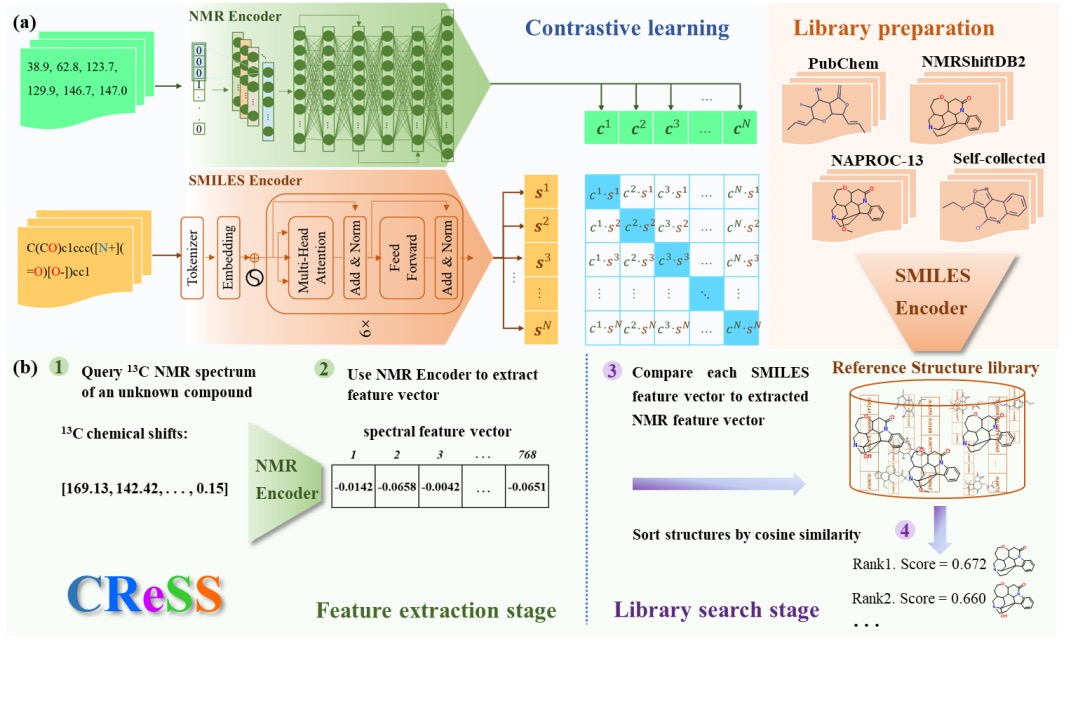
图 7. 跨模态碳谱-结构检索方法。(a)对比学习 (2)CReSS 流程
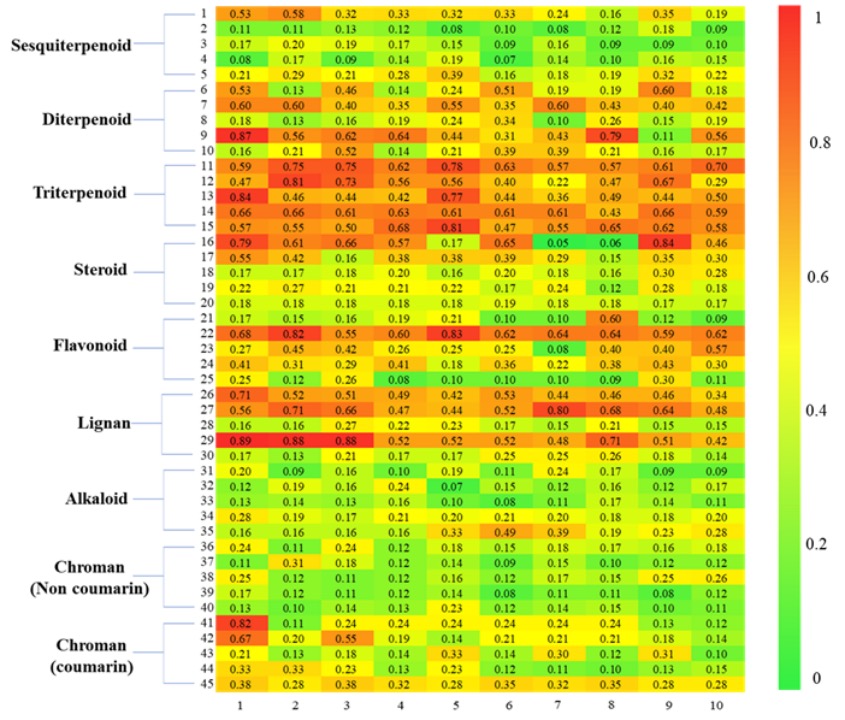
图 8. “新化合物”的正确结构与 CReSS 检索的候选结构之间的 Tanimoto 相似度热图
(2) CMGNet
CMGNet(Conditional Molecular Generation Net)是一个基于双向自回归 Transformer(BART)的条件生成模型,CMGNet 在 3.6 亿分子组成的数据集上进行了大规模预训练从而充分学习理解了分子结构,然后对两个不同粒度级别的 NMR 数据集进行微调,以适应下游的结构解析任务。与 CReSS 依赖于在分子结构库进行检索的方式不同,CMGNet 不需要任何底库,直接由模型生成分子结构,而且除了可以接受碳谱数据作为输入,还能够接受专家推断的分子式和分子片段作为额外输入以辅助解析结构,特别适用于分子量大和结构复杂的分子。CMGNet 仅根据未知化合物的 13C NMR 化学位移数据就能生成分子结构,当加上分子式和片段信息时,模型的 Recovery@10 能达到 94.17%,即 94.17%的化合物都能在模型生成的前 10 名候选结构中找到正确的。此外,CMGNet 在各类化合物的结构解析任务和结构纠错任务中表现良好。CMGNet 学习了分子的固有性质和专家的先验知识,对结构-性质关系有了更深刻的理解,为深度学习辅助逆问题解决的新范式铺平了道路。
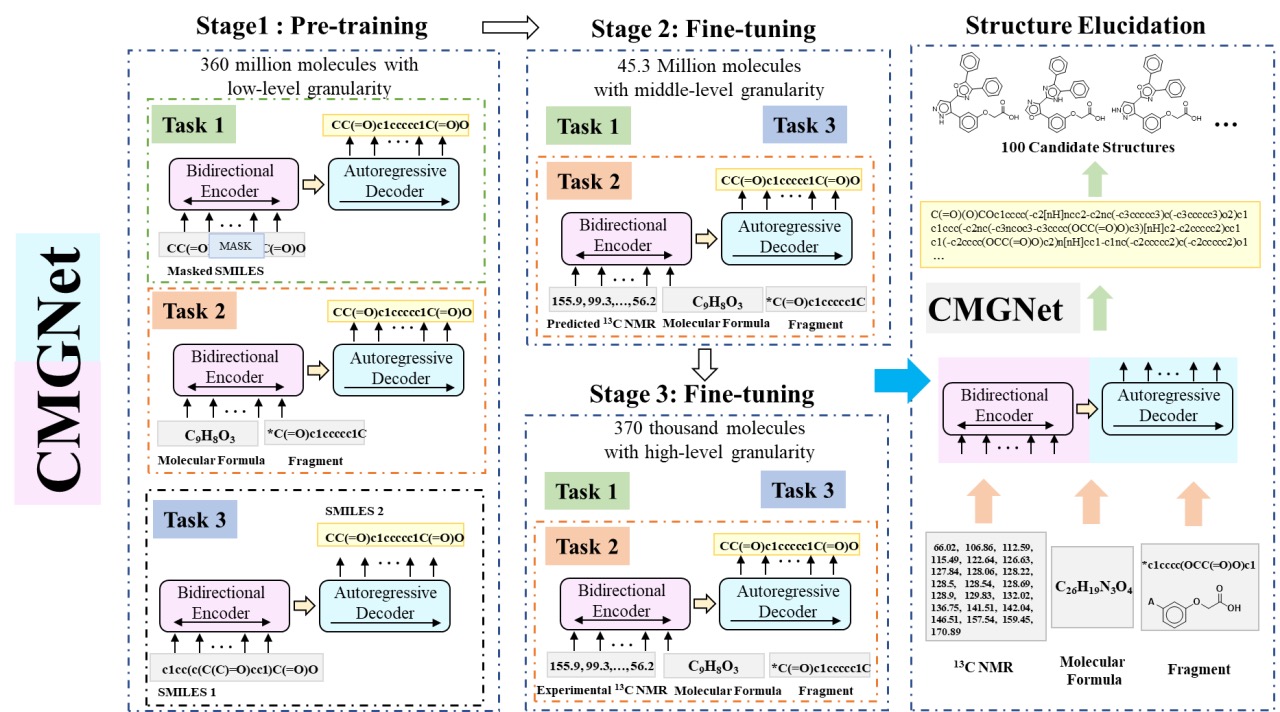
图 9. CMGNet 的训练和推理流程。
表 1. CMGNet 在不同附加测试集约束下的性能比较。
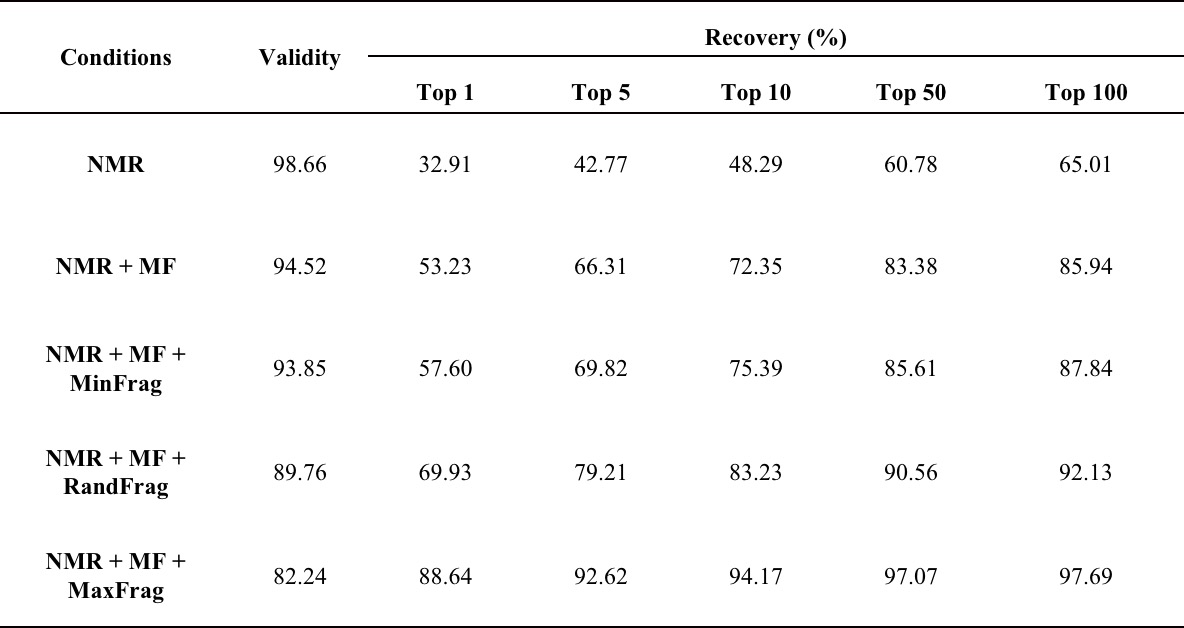
说明:MF-Molecular Formula, MinFrag-the Minimum Fragment, RandFrag-Random Fragment, MaxFrag-the Maximum Fragment.
5. 相关文献
[1] Cross-Modal Retrieval between (13)C Nmr Spectra and Structures for Compound Identification Using Deep Contrastive Learning. Yang, Z.; Song, J.; Yang, M., et al. Anal. Chem. 2021, 93, 16947−16955. DOI: 10.1021/acs.analchem.1c04307
[2] Conditional Molecular Generation Net Enables Automated Structure Elucidation Based on 13C NMR Spectra and Prior Knowledge. Yao L., Yang M., Song J., et al. Anal Chem. 2023 Mar 28;95(12):5393-5401. doi: 10.1021/acs.analchem.2c05817
