分子工厂
1. 分子工厂概述
分子工厂是一种全流程的分子设计方法。
如何设计出结构新颖且有治疗效果的可成药分子一直是新药研发的核心所在。与人类相比,以深度学习为代表的 AI 分子生成模型拥有从大量数据中学习的能力,能够探索更广阔的化学空间并具备针对特定分子性质的分子优化能力。当前 AI 分子生成技术,如骨架跃迁技术、片段生成技术以及从头设计技术已被广泛应用于许多重要的分子设计和分子优化项目当中,它们能更有效地遍历化学空间,使得研究人员需要考虑的分子数量聚焦在针对特定靶点的一个更小的范围内。除此之外,在通过 AI 分子生成模型针对给定靶点和性质约束条件获得大量的分子之后,搭建完善且高效的自动化筛选流程,以基于分子的物理化学性质和成药性、分子与靶点的关键相互作用以及结合药化专家的经验筛选出潜在有效的分子,也是提高研发效率的关键所在。
通过将 AI 分子生成技术和自动化筛选流程进行高效整合,我们构建了分子工厂模块,用于协助研究人员和相关从业者开展高效的药物研发流程。
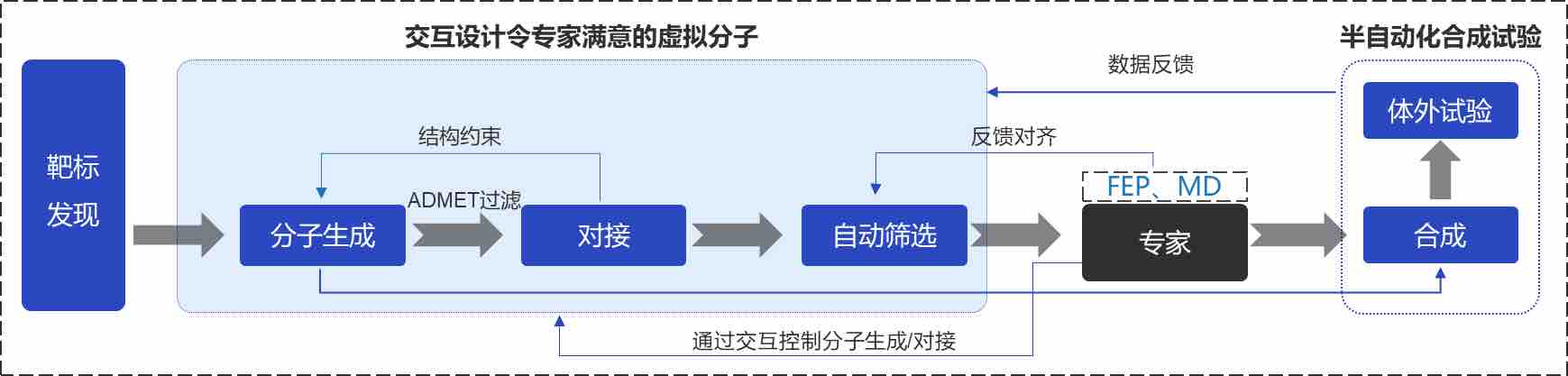
图 1. 分子工厂的核心设计理念
用户在分析特定蛋白质靶点确定分子生成方案以及分子筛选条件后,便可利用分子工厂模块自动完成包括基于分子生成模型获取待筛选分子和基于给定的分子筛选条件对待筛选分子进行筛选获取最终的潜在活性分子在内的整个流程。分子工厂的核心特色主要包括:
(1)高度自动化的分子设计筛选全流程:分子工厂涵盖了包括分子生成、物理化学性质筛选、分子成药性筛选、分子对接、蛋白质-配体关键相互作用筛选在内的药物分子设计经典流程。其中蛋白质-配体关键相互作用筛选在传统上是极为耗时耗力的环节,往往由人工查看。分子工厂将该环节自动化,对分子对接得到的复合物构象进行批量相互作用分析,并根据专家指定的关键子结构和关键相互作用开展自动过滤。
(2)分子生成模型覆盖功能齐全:分子生成模型同时支持基于配体的药物设计和基于结构的药物设计,因此用户可以自由选择是否上传蛋白质结构。分子设计模式涵盖了 R-基生成、Linker 生成、骨架跃迁和从头设计等多种分子设计模式,能够满足多种设计需求。不仅如此,分子工厂也支持用户上传靶点已知的活性分子数据集,从而自动对分子生成模型进行微调,以进一步提升针对特定靶点进行分子生成的特异性。
(3)应用了当前所有半柔性对接方法中精度最高的分子对接模型 CarsiDock:分子工厂整合了碳硅智慧自主开发的 AI 对接模型 CarsiDock,开发过程中利用了物理计算产生的海量复合物结构模拟数据,能够快速精确预测配体分子和蛋白质的结合构象。
(4)与专家经验一致的筛选条件:分子工厂建模了专家的分子设计经验,对经分子生成模型获取的候选分子自动开展专家经验过滤,包括不合理子结构、分子量、拓扑极性表面积、脂溶性、水溶性、PAINS、Alerts、定量评估类药性、氢键供体数目、氢键受体数目、可合成性、可旋转键数目、环数目、螺环数目、桥环数目和大环数目等方面的过滤。其中不合理子结构是类药分子中不常见的子结构,包括稳定性低的子结构、罕见的子结构和合成难度高的子结构等多种类别,均来自资深药物化学专家的经验总结。
(5)大幅提升开发效率:基于分子工厂开展药物设计和筛选,可在一周内完成一轮分子优化,降低 50%早期研发时间,降低 70%早期研发成本。
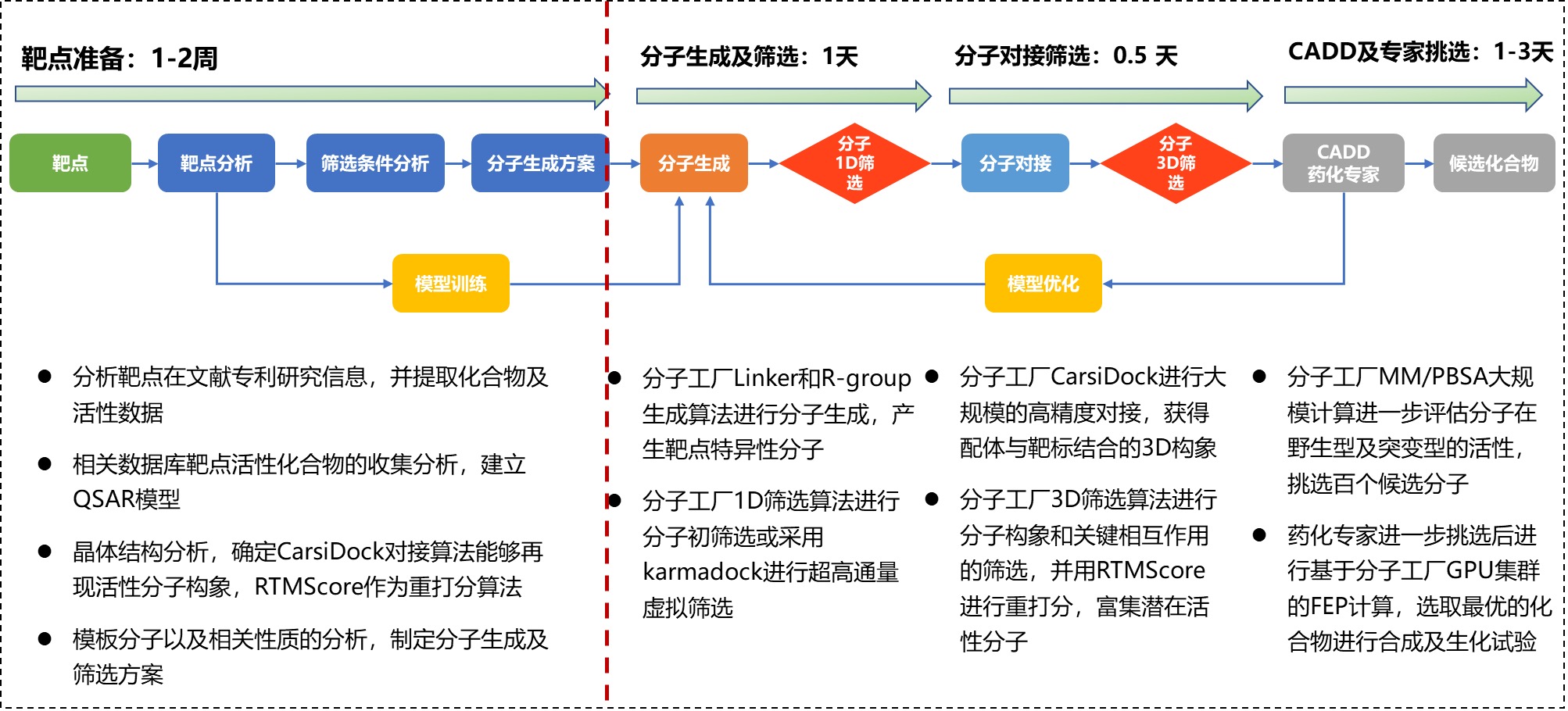
图 2. 分子工厂辅助药物设计的流程和时间耗费
2. 使用说明
为完成全流程的自动分子设计,用户需要依次设置:数据选择-筛选条件配置-任务命名(可忽略)-提交任务(必点)。
(1) 数据选择
数据选择用于配置分子生成相关参数。
- 生成类型
目前平台支持 5 种分子生成类型,分别为:
R-Group:在参考分子的基础上,取代一个 R 基
Sidechain-base:在参考分子的基础上,取代多个 R 基
Linker-based:在参考分子的基础上,取代中间的 Linker 保留两端的结构
Scaffold-hopping:在参考分子的基础上,取代中间的骨架,保留末端的多个结构
De Novo:从头生成
- 上传蛋白及设置口袋(可选项)
如果用户在分子生成时不需要基于蛋白质结构,可以关闭跳过这一步;否则,如果希望基于蛋白结构生成分子,则需要上传蛋白并设置口袋。
- 上传蛋白
平台提供了两种上传蛋白的方式:上传文件和数据中心;蛋白上传后,左边将自动可视化显示蛋白。
从本地上传文件:复选框选中“上传文件”,通过点击下方按钮选择本地文件即可。当前支持的文件格式为.pdb,选择完文件以后,该文件名称将显示在按钮上。
从数据中心选择:复选框选中“数据中心”,通过点击下方按钮页面出现弹窗,点击文件名称来选择数据中心的数据,点击完之后弹窗消失。
- 设置口袋
指定分子的结合口袋,这里支持两种方式:根据您上传的 PDB 文件,系统会自动选择分子质量最大的分子作为口袋,并根据该配体分子来确定几何中心坐标和 Box Size 大小;您也可以自定义口袋,手动调整口袋的 Coordinate 和 Box Size 。
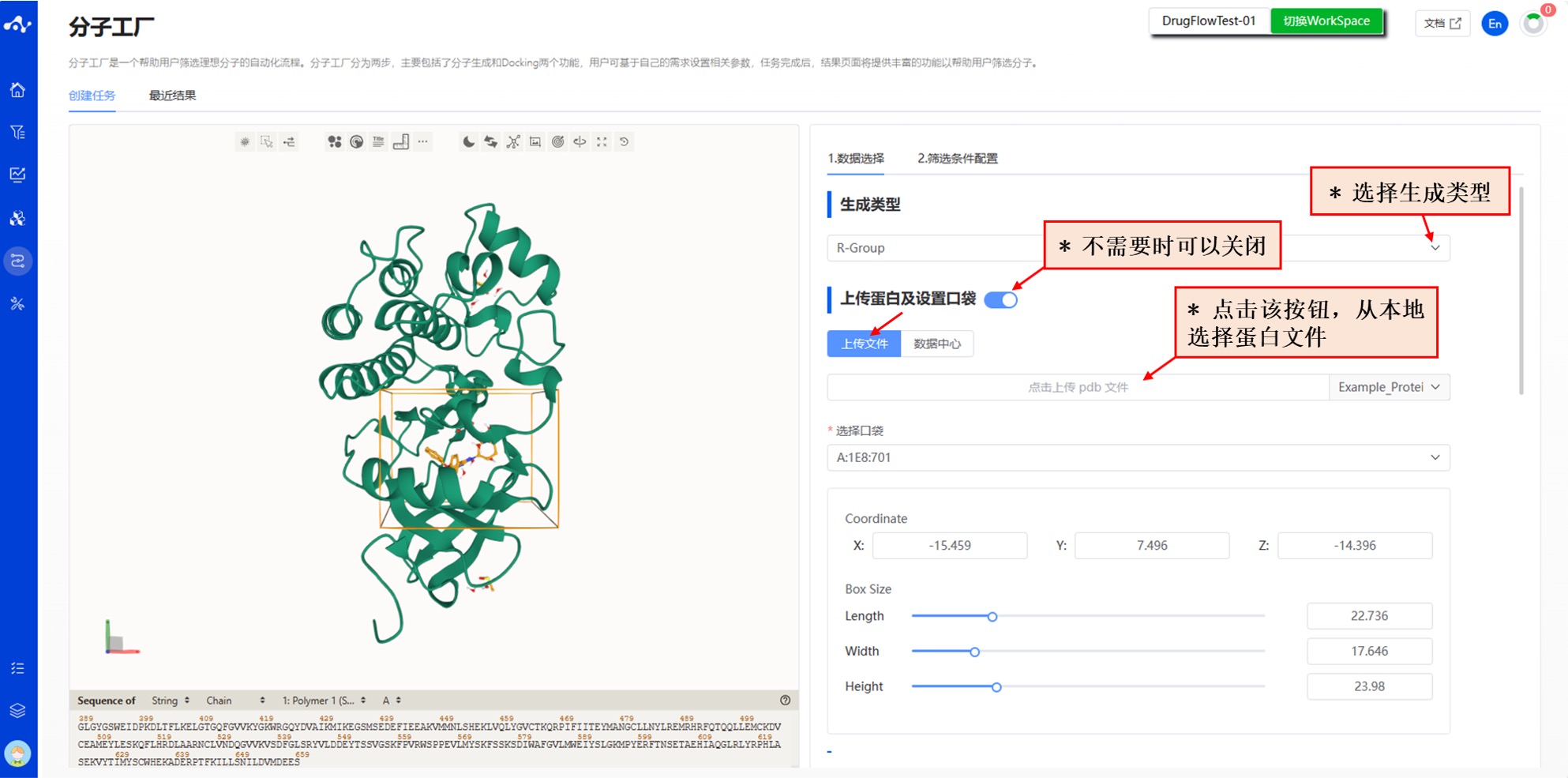
图 3. 分子工厂任务创建页面——上传蛋白及设置口袋
- 上传并编辑参考分子/参考片段
上传参考分子/参考片段,平台支持 4 种上传方式:使用复合物配体、输入 SMILES 字符串、从本地上传文件(.sdf)和从数据中心选择。
使用分子编辑器从上传的参考分子/参考片段中选择并删除期望被替代掉的子结构,平台自动根据编辑结果识别分子生长的起始原子位置。
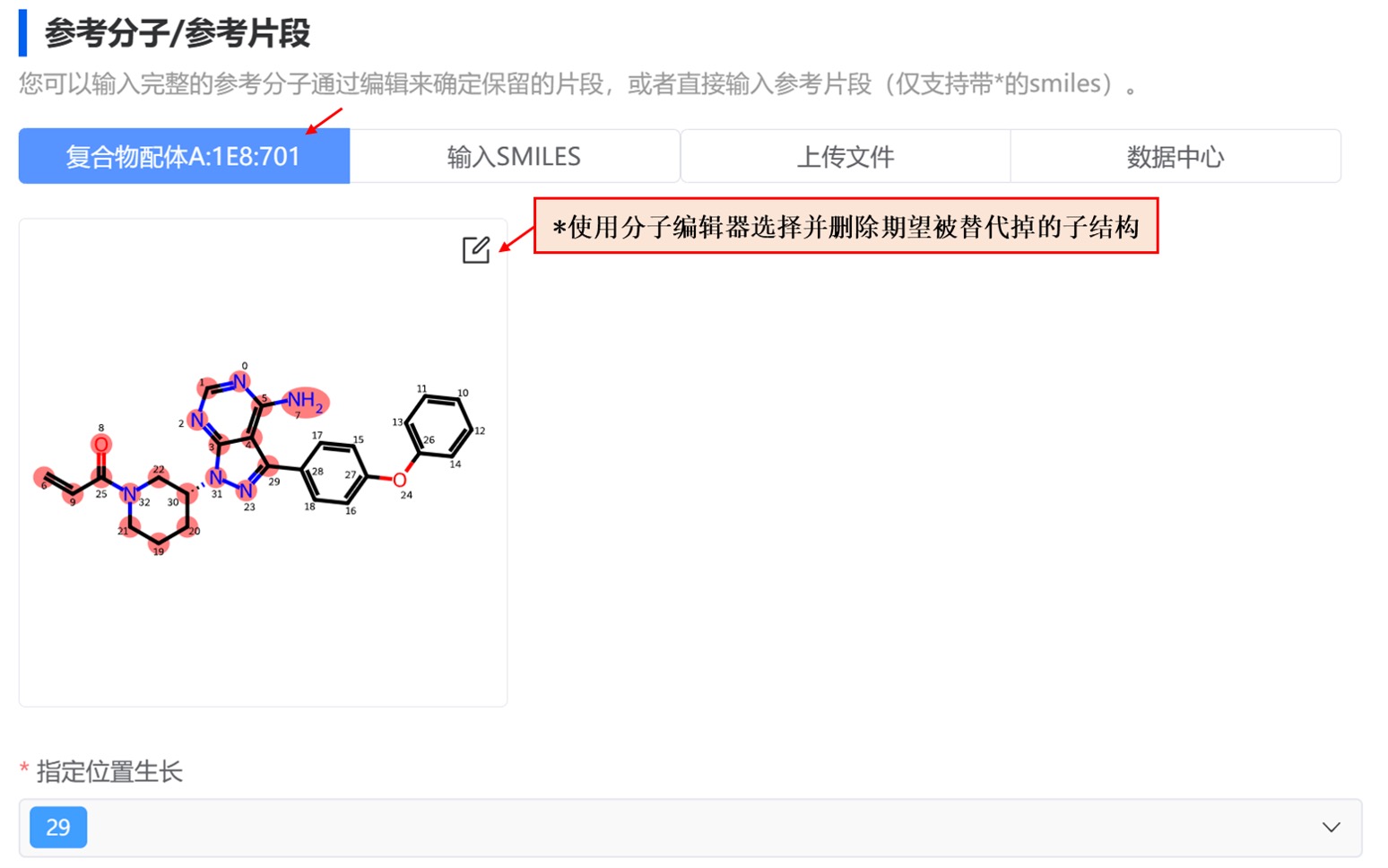
图 4. 上传并编辑参考分子/参考片段
- 上传活性分子(可选项)
如果用户已经收集了一批靶点对应的活性分子,可以上传活性分子,平台将会自动利用这批活性分子对生成模型进行微调,并基于微调后的模型生成分子,以使得生成的分子在结构/性质上与活性分子更相近。如果不需要的话也可以关闭跳过这一步。
平台提供了两种上传分子的方式:上传文件和数据中心
从本地上传文件:复选框选中“上传文件”,通过点击下方按钮选择本地文件即可。选择完文件以后,该文件名称将显示在按钮上。
从数据中心选择:复选框选中“数据中心”,通过点击下方按钮页面出现弹窗,点击文件名称来选择数据中心的数据,点击完之后弹窗消失。
当前支持的文件格式: .sdf/.csv;如上传 csv 文件,则需指定 smiles 列;如上传 sdf 文件,则不需要指定。
为保证良好的微调效果,所上传活性分子的数目应不低于 300。
- 设置生成方法及结束条件
平台提供了多种分子生成方法:ResGen, CarsiLinker, CarsiLinker3D, FragGPT, Delete, Smiles-GPT,每种方法支持的分子生成模式不完全相同,系统会根据您的输入自动确定生成方法,您可以在取消某个可用的算法。
设置生成数量和生成时间,一旦达到生成数量或生成时间中的任一条件,平台会自动终止生成任务。
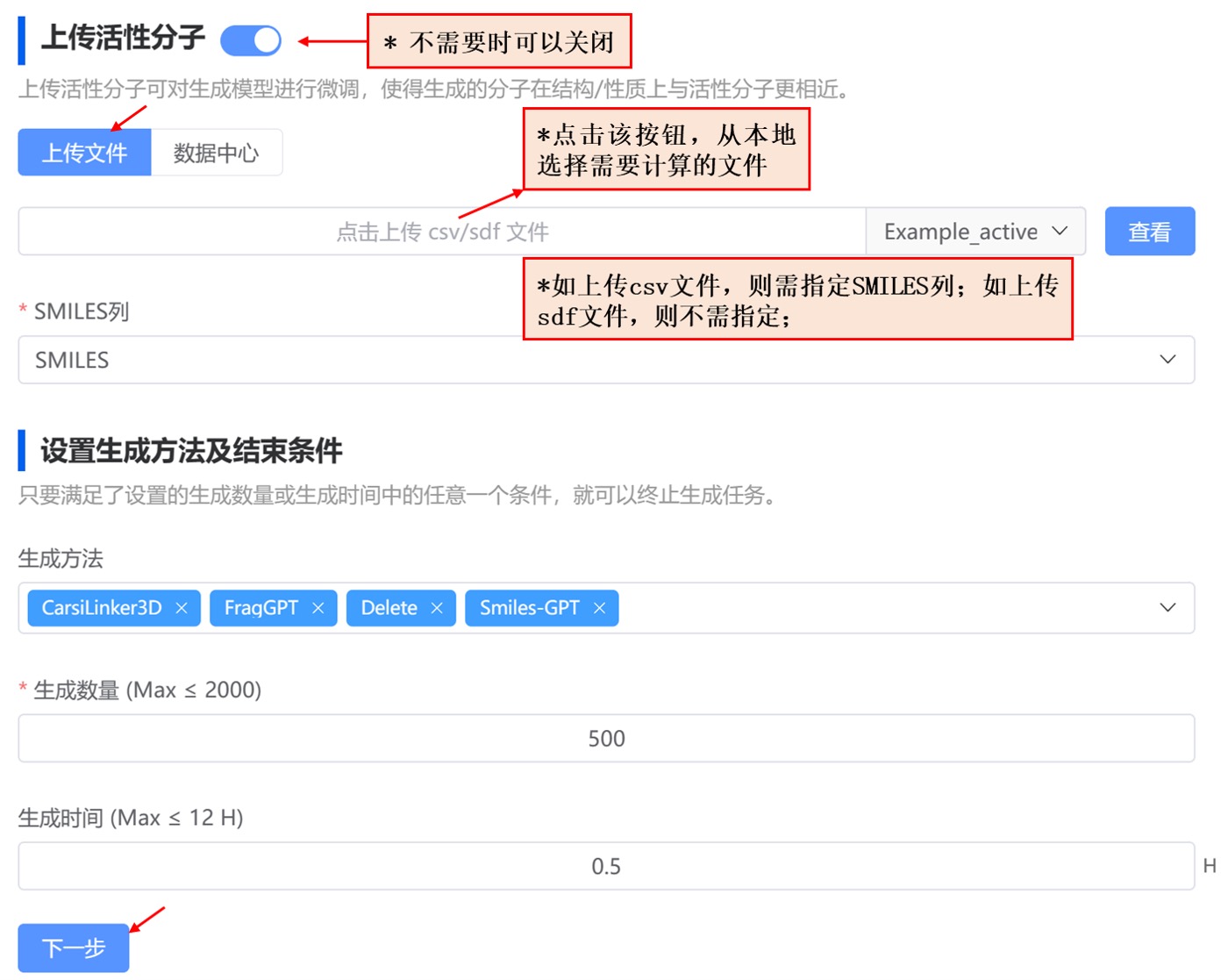
图 5. 上传活性分子和设置生成方法/结束条件
在确定好所有的参数后,点击下一步,进入到筛选参数配置。
(2) 筛选条件配置
筛选条件配置用于配置分子筛选条件相关参数。
- 2D 属性过滤(可选项)
如果用户不希望对生成的分子进行 2D 属性过滤,可以关闭跳过这一步;如果希望对生成的分子进行 2D 属性过滤,则继续选择属性种类,并调整属性的目标范围。平台支持 4 种属性,包括 MW、TPSA、LogP 和 LogS。
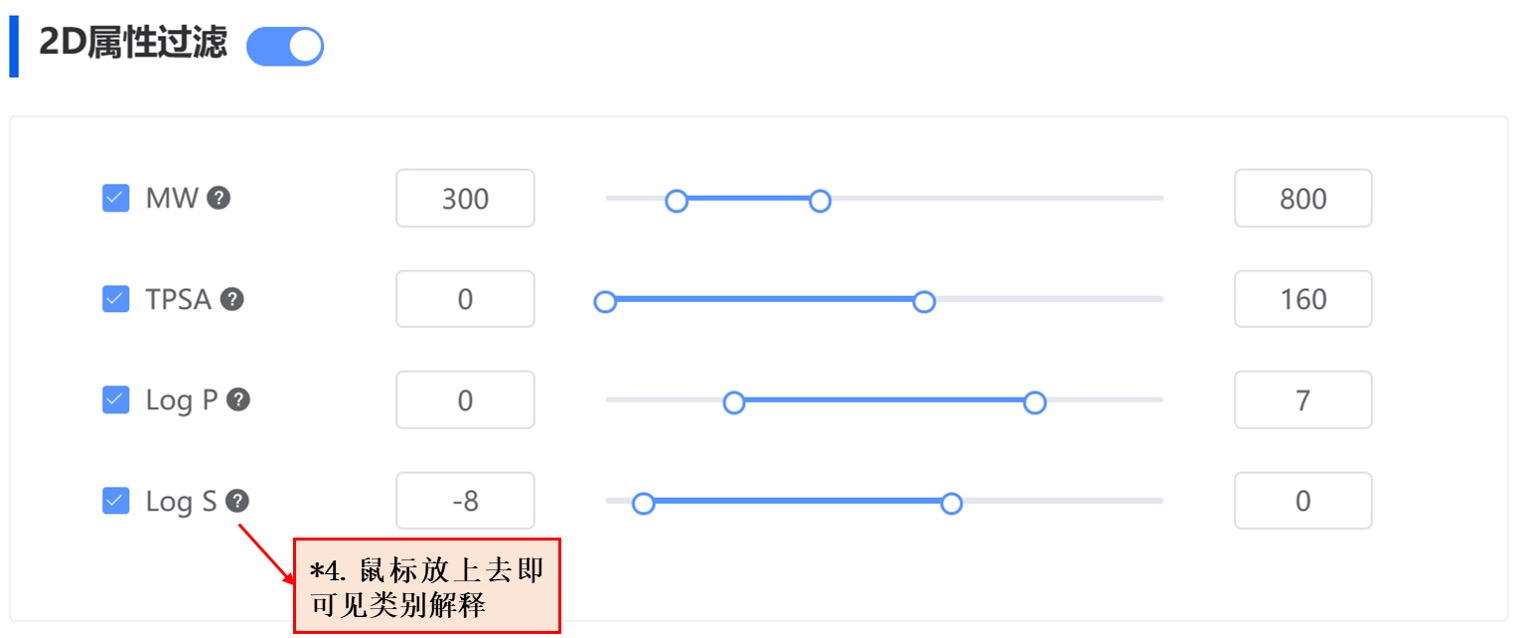
图 6. 筛选条件配置——2D 属性过滤
- Docking 相关参数设置(可选项)
这一步用于对生成的分子进行自动对接以生成复合物构象,如果用户不需要这一步,也可以选择跳过。如果需要,则继续依次配置生成异构体参数,并从 CarsiDock 和 AutoDock Vina 中选择一种对接方法。
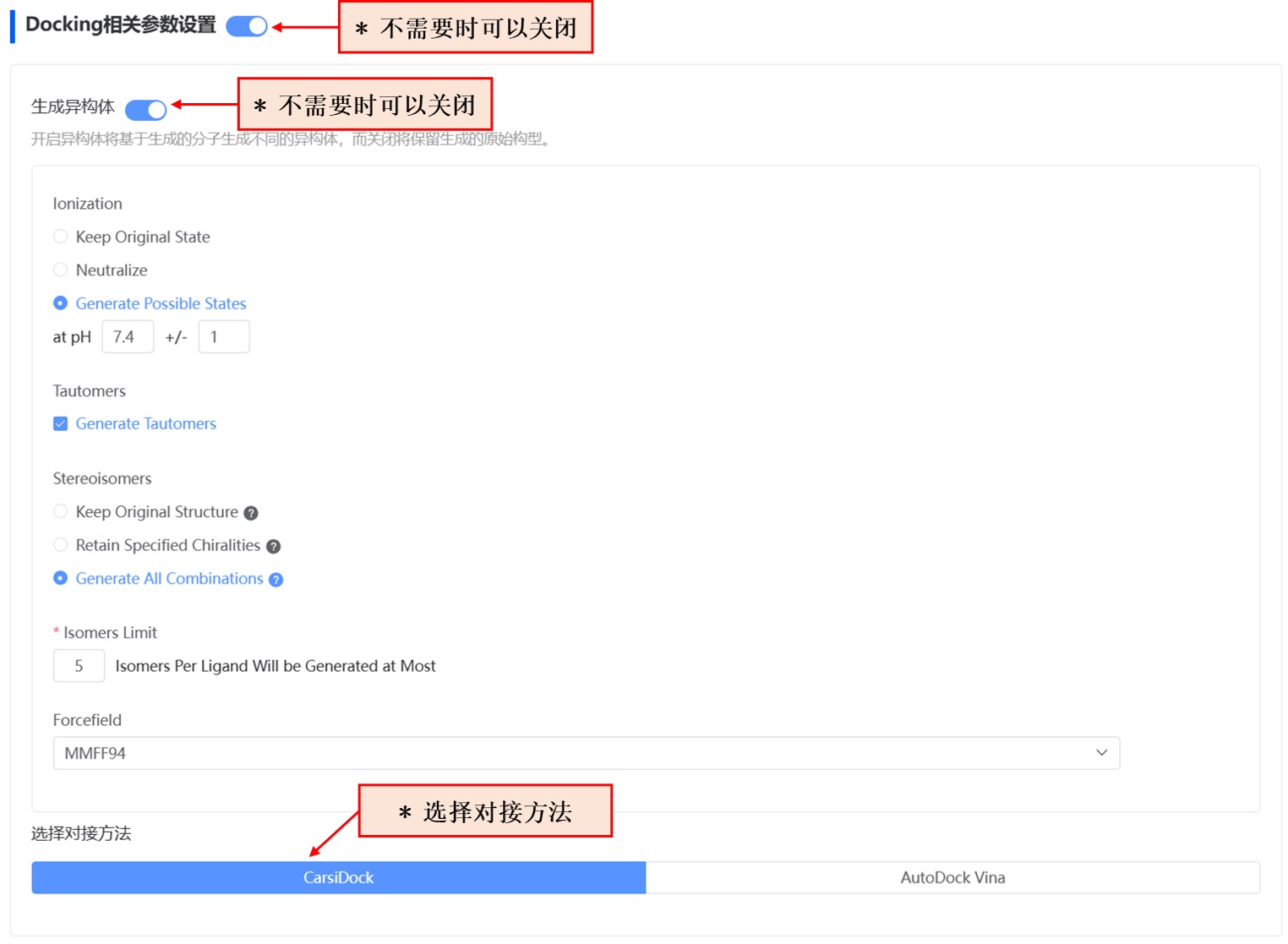
图 7. 筛选条件配置——Docking 相关参数设置
- 对接构象比较(可选项)
这一步用于比较生成分子和参考分子与蛋白质结合时的公共子结构之间 3D 构象差异。如果不需要这一步,也可以选择跳过。如果需要,则继续使用分子编辑器选择并删除非公共子结构的结构区域。
- 原子空间位置检测(可选项)
这一步用于指定参考分子中的关键原子,以检测在复合物构象中被指定原子的 5 埃范围内,生成的分子中是否存在相同类型的原子。如果不需要这一步,也可以选择跳过。如果需要,则从下拉框选择原子。
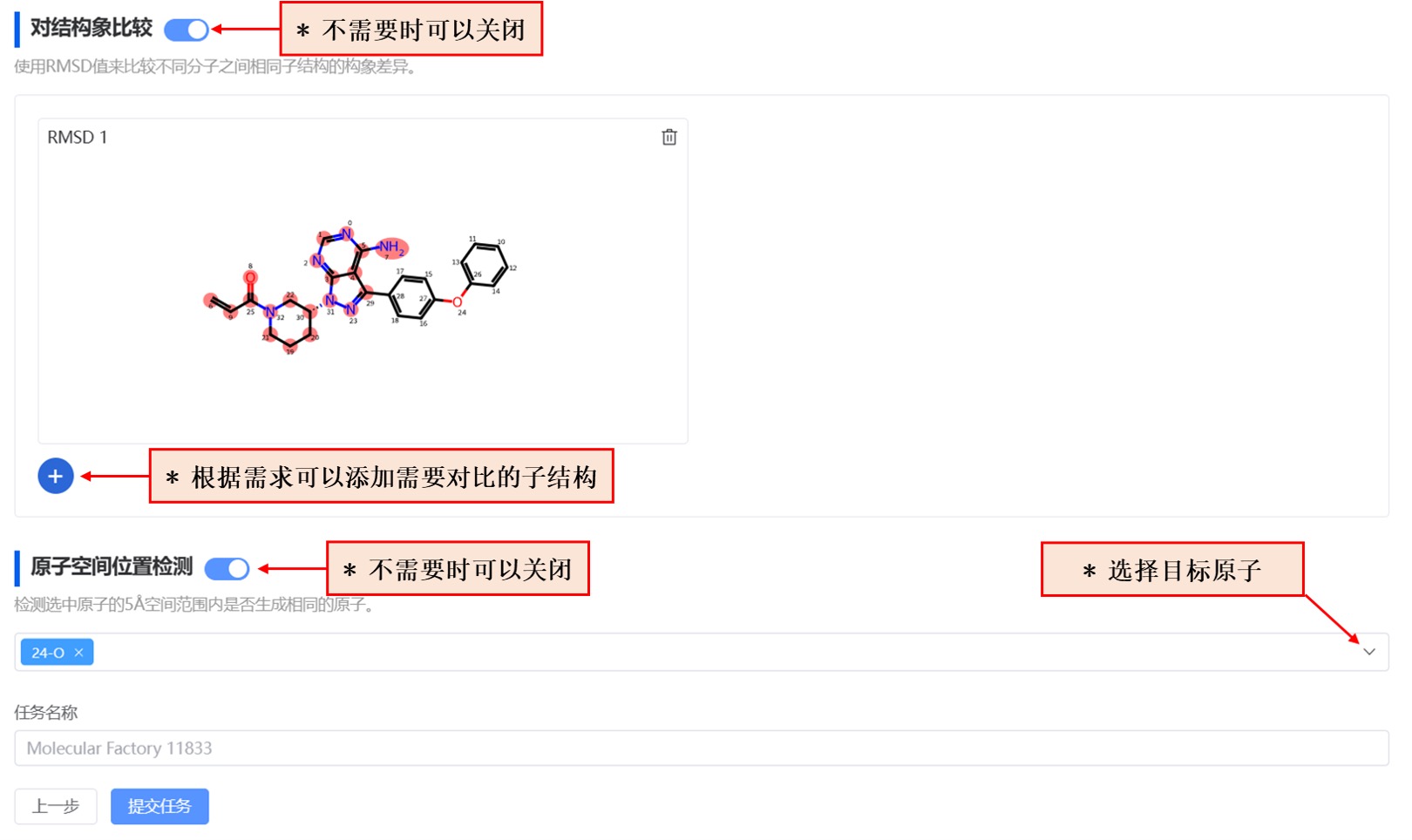
图 8. 对接构象比较和原子空间位置检测
在确定好所有的参数后,命名任务名称,点击提交即完成任务提交操作。
(3) 运行进度和结果查看
提交任务后,页面会自动跳入当前页面的“最近结果”子页面中,您可以在该页面查看当前模块的任务运行状态(进度条),也可在右上角的“运行中“下拉框中查看所有模块正在运行的任务。当任务结束后,即可点击“结果详情”按钮进入结果页面。
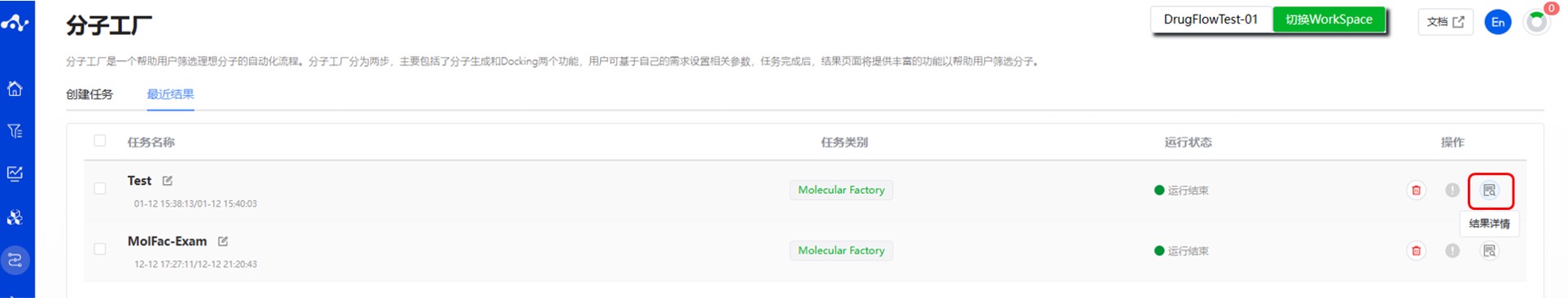
图 9. 查看结果
3. 结果分析
结果页面由顶部的 Summary、左边的分子列表和右边的蛋白可视化区域组成。默认状态下,左边的结果详情区展示的是网格页面,可以切换至列表和聚类子页面。其中,网格子页面提供了一个简洁的查看分子结构的页面,而列表页面则提供了详细的计算结果,方便你分析当前的数据,而聚类子页面则将得到的分子按照 Murcko 骨架对分子进行分类展示。 蛋白可视化区域是一个固定的内容,无论左边在什么子页面,蛋白可视化区域都将展示蛋白-配体的对接模式,用户可通过右下角的“上一个”、“下一个”快速浏览分子间的相互作用。
网格页面下,每个分子卡片上展示了分子的结构,卡片右上角的三个符号分别对应于三种操作,即收藏、不喜欢和结果详情,用户可以利用收藏和不喜欢对分子进行标记,点击结果详情则会跳转到跳转到分子详情页。
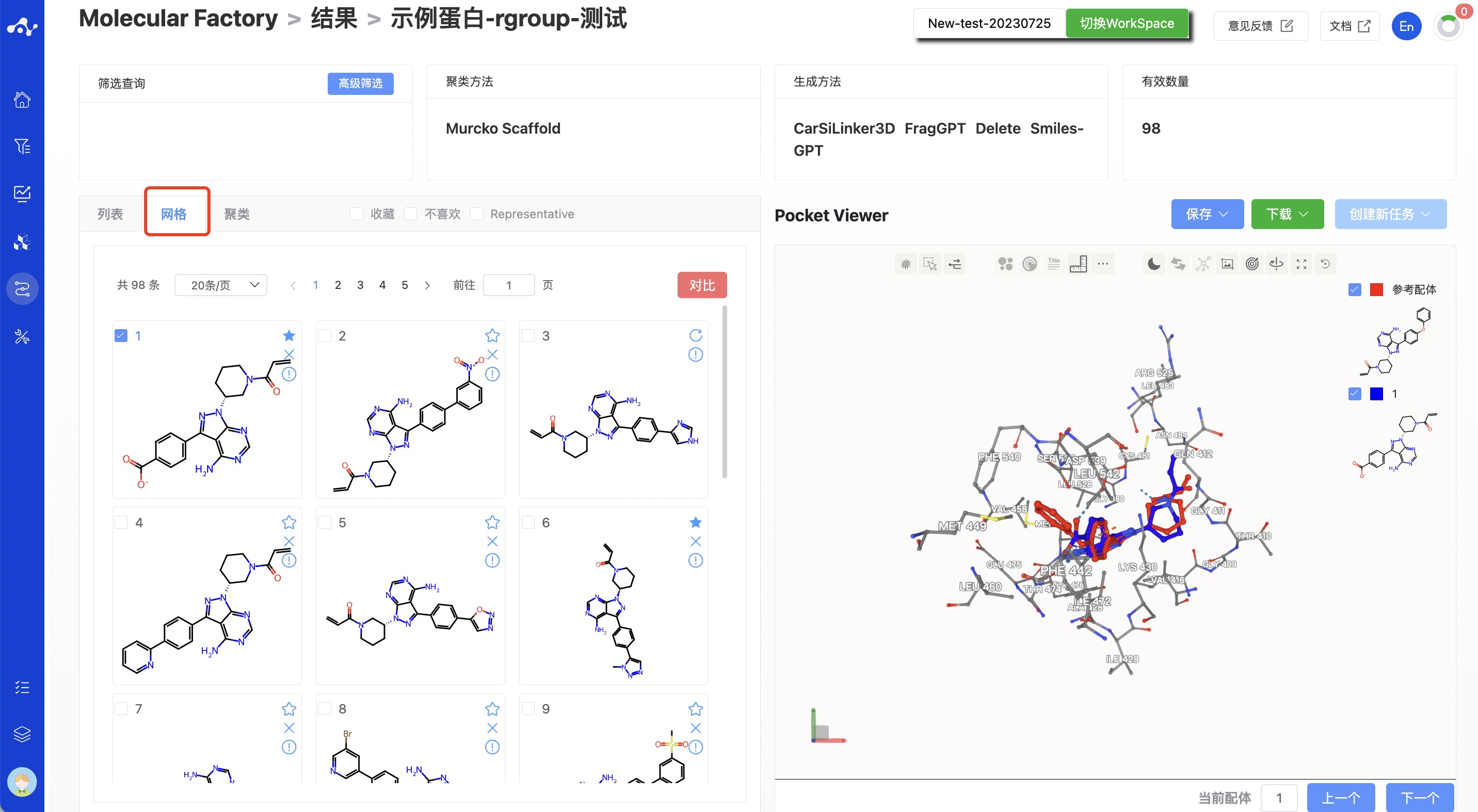
图 10. 分子工厂结果页面-网格页面
列表页面下,可以详细的查看分子的信息,包含生成分子的 ID、操作、结构和多项性质。把鼠标移入每个性质的名称上,可查看该性质对应的解释。点击列表中的类别名字可重新排序,点击一次为升序,再点一次为降序,再点一次即恢复原始排序。
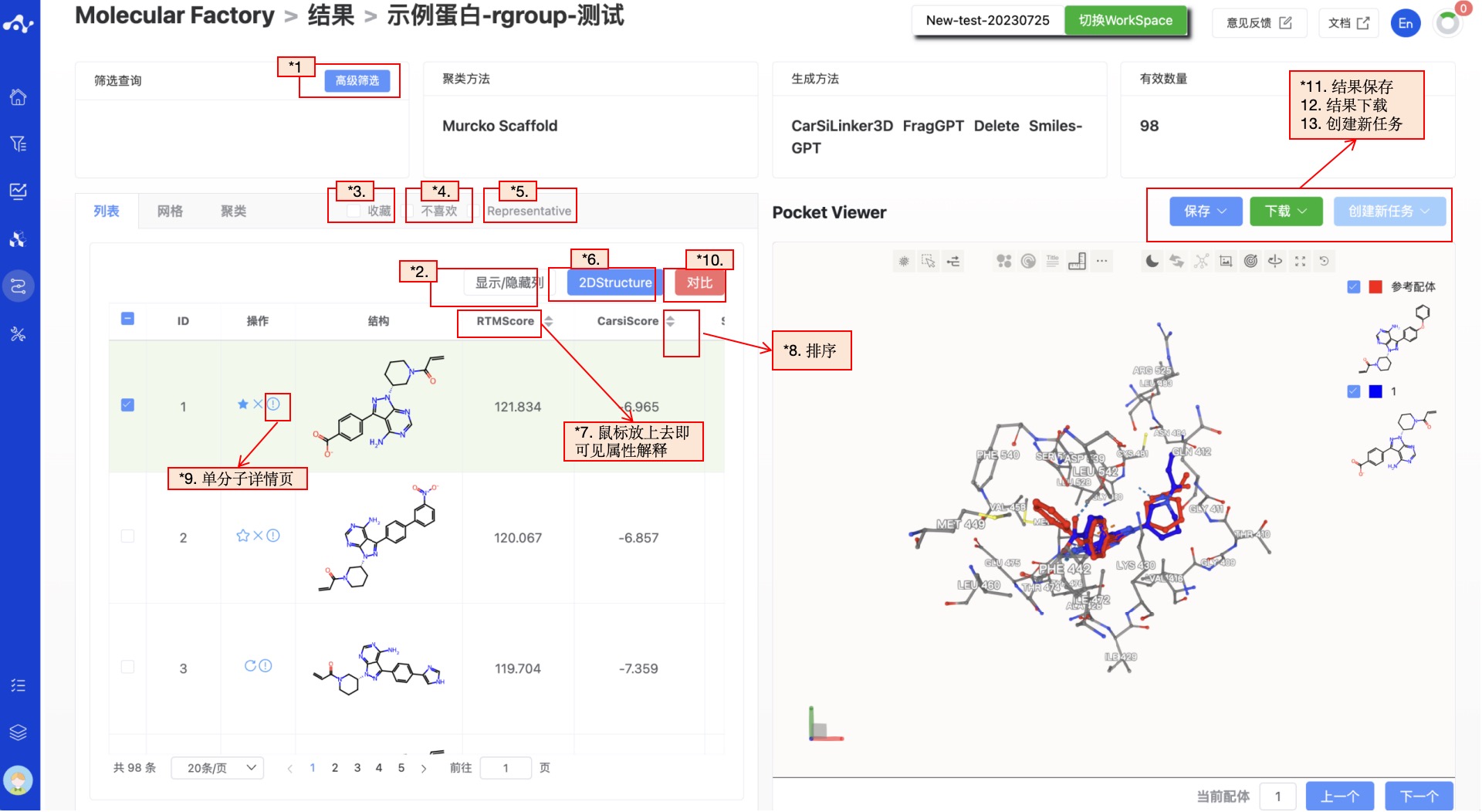
图 11. 分子工厂结果页面-列表页面
聚类页面下,根据 Murcko Scaffold 对生成的分子进行聚类显示,同一个骨架将会放在一起展示。
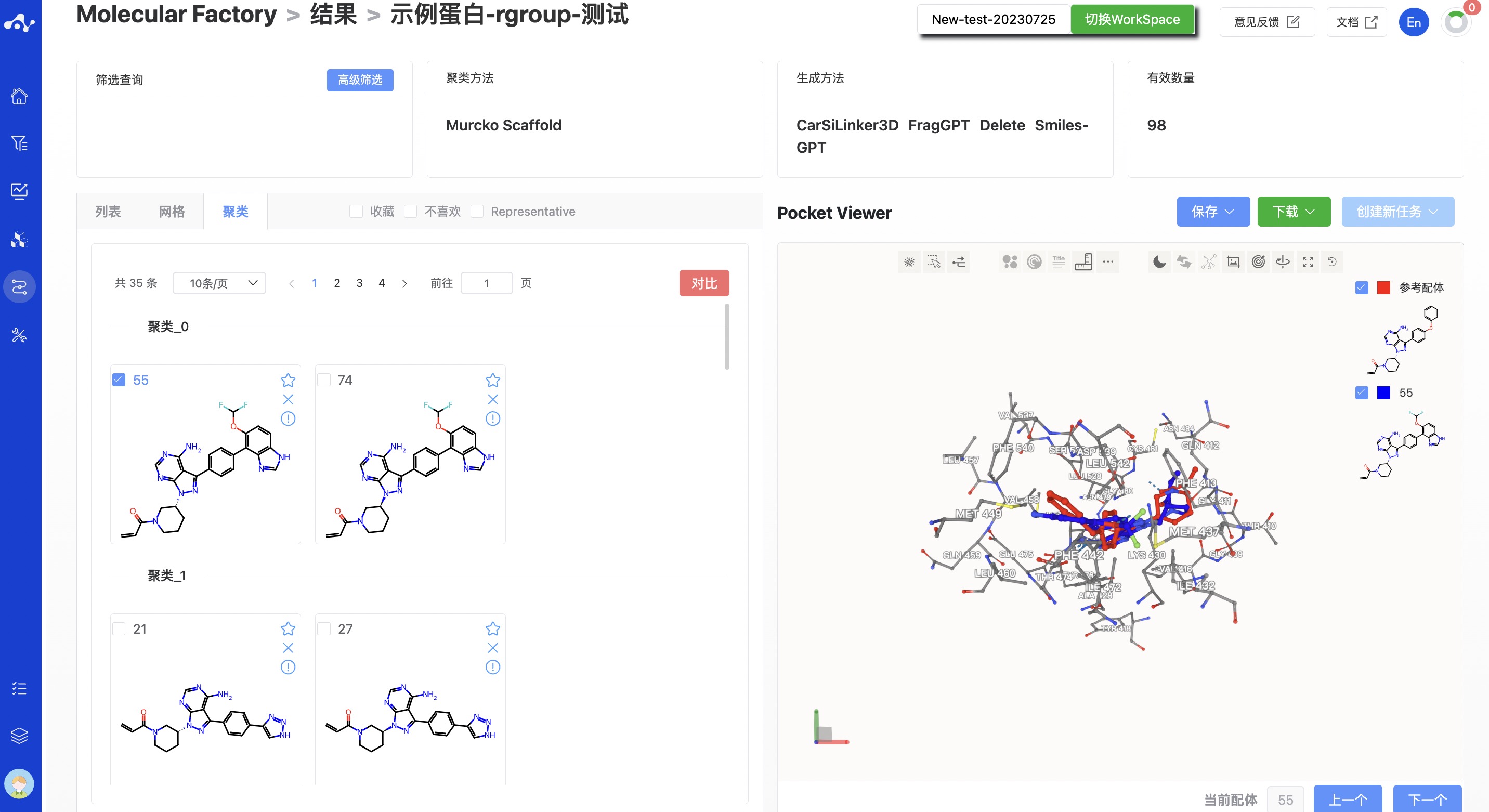
图 12. 分子工厂结果页面-聚类页面
(1) 高级筛选
高级筛选提供了范围筛选,可进一步筛选出某性质指定范围内的分子,以排除不符合预期结果的分子。进行高级筛选后,页面上只会显示符合过滤条件的分子。
(2) 显示/隐藏列
默认的结果列表展示所有的信息。当你不想显示该性质时,取消该性质的选中即可,左边的结果列表将根据控制栏中的选择进行实时显示。
(3) 收藏
该功能主要用于帮助用户标记喜欢的分子。当你点击收藏某个分子后,该图标将会被点亮,意味着该分子被标记为收藏分子。点击收藏的勾选框之后,页面将只会显示被收藏的分子。点击收藏以后,可以再次点击该图表取消收藏。
(4) 不喜欢
该功能主要用于帮助用户标记不喜欢的分子。当点击不喜欢某个分子后,收藏的图标将消失,且不喜欢的图标会改成恢复图标,意味着用户可以随时修改该分子的标签。点击不喜欢的勾选框之后,页面将只会显示被标记为不喜欢的分子。
(5)Representative
在列表、网格和聚类这 3 个选项右侧,还有另外 3 个选项,分别是收藏、不喜欢和 Representative。点击收藏,则结果详情区只展示被用户标记为收藏的分子。点击 Representative,则结果详情区只展示聚类后每个类别中最具代表性的分子。
(6) 2D Structure
默认该按钮被选中,列表中正常显示配体结构,点击一次后可取消选中,则列表中该分子结构被隐藏。
(7) 属性解释
把鼠标移入每个性质的名称上,可查看该属性对应的解释。
(8) 排序
点击结果列表中的性质名字可重新排序,如 CarsiScore,点击一次为升序,再点一次为降序,再点一次即恢复原始排序。
(9) 单个分子详情页
点击“分子详情”按钮,页面将跳转至单分子详情页,该页面全面展示了分子的各种信息让用户快速了解该分子的详细信息,包括此次计算的内容,以及该分子的 Inno-ADMET 属性和对接构象。
- 点击 Inno-ADMET,可以看到当前分子的物化性质、药化性质、类药性、吸收、分布、代谢、排泄、和毒性等性质。将鼠标放在各项性质右侧,可以查看释义及推荐范围;
- 点击对接构象,可以看到当前分子与蛋白口袋结合时的的复合物构象。另外,也提供了参考分子的结合构象便于比较。
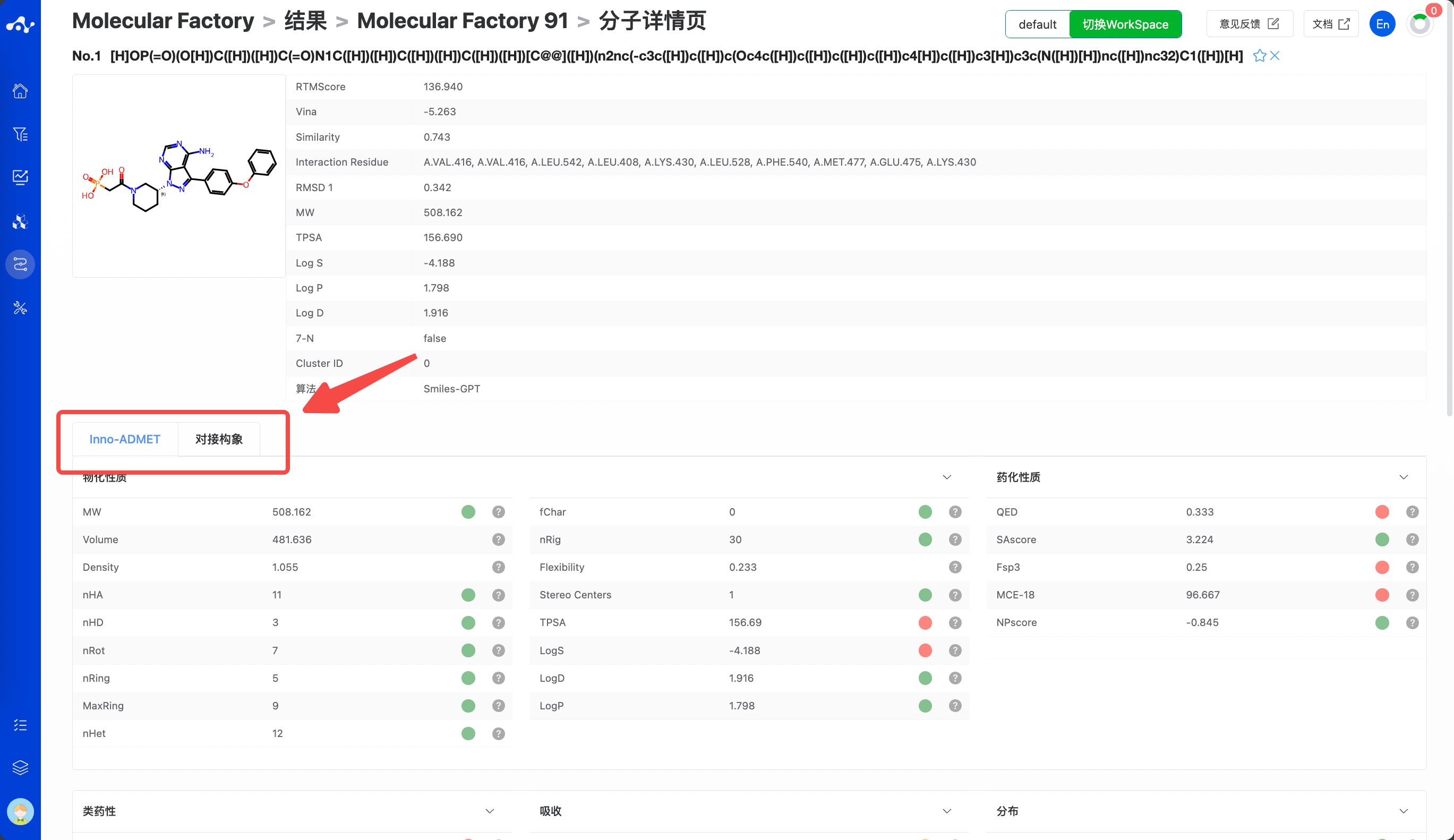
图 13. 单分子详情页
(10) 对比
每个分子的序号前面都有一个复选框,当你同时勾选多个分子时,即可点击【对比】按钮,页面即跳转到对比页面,方便进行多个分子的全面比较,当你有参考分子时,则会与参考分子进行对比。该页面与单分子详情页比较类似,包含了该分子的 Inno-ADMET 属性、ChemFH 属性、Inno-SA 属性和对接构象。
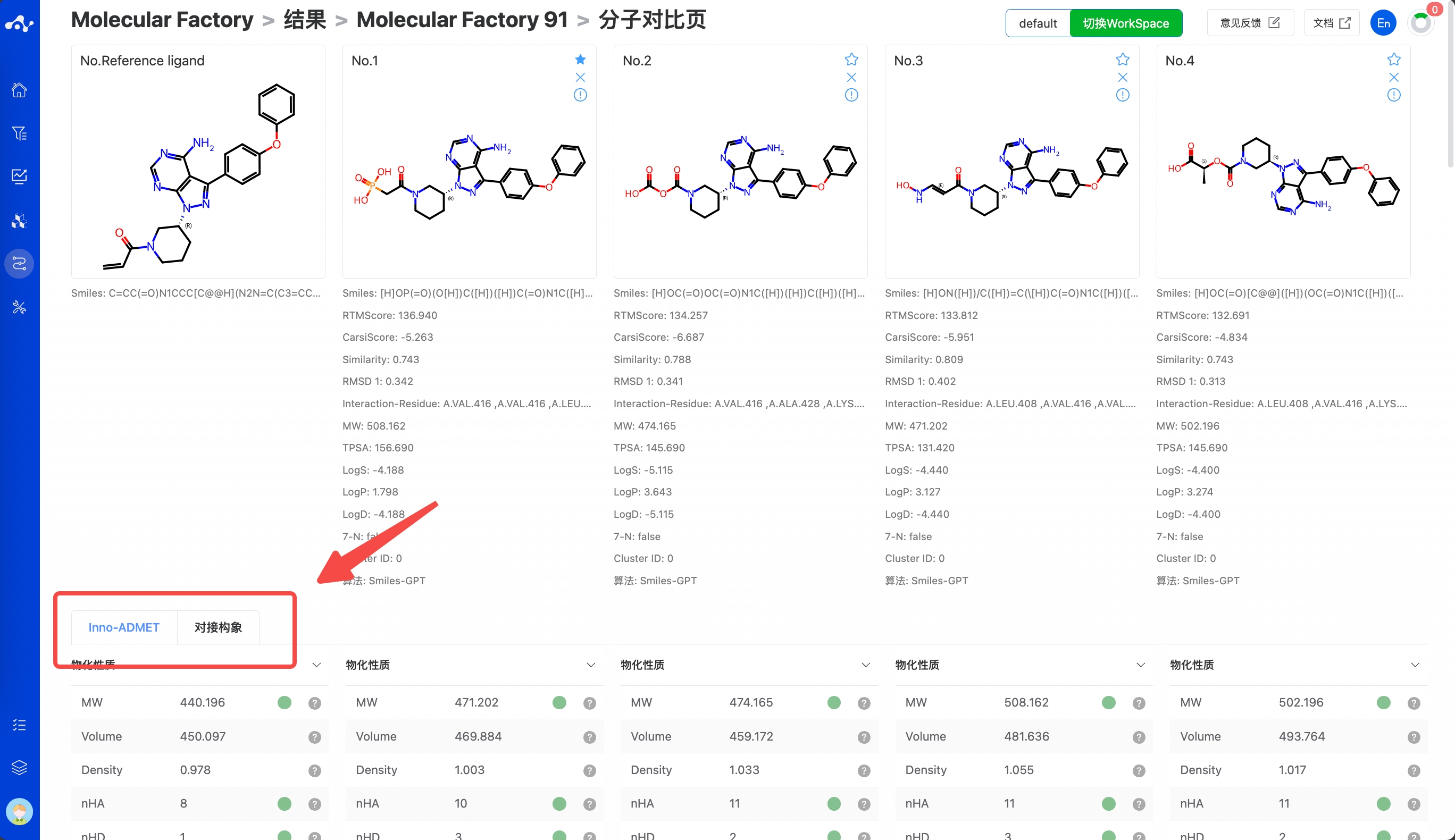
图 14. 分子对比页
(11) 保存
点击“保存”,系统将弹出下拉框让你选择保存的文件格式(目前仅支持.csv/.sdf)。确定好保存的文件样式后,保存相应的数据为 sdf 或 csv 文件至数据中心。保存的内容是页面展示的有效数量的分子,而这些分子通常是根据你的显示隐藏列的条件、高级筛选的条件、收藏或者是不喜欢来获取的。
(12)下载
点击“下载”,系统将弹出下拉框让你选择下载的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将下载相应的数据为 sdf 或 csv 文件至本地设备上。下载的内容与保存的方式一致,也是下载的页面展示的有效数量的分子,而这些分子通常是根据你的显示隐藏列的条件、高级筛选的条件、收藏或者是不喜欢来获取的。
(13) 创建新任务
创建新任务的前提是先保存数据为一个文件,在未执行保存操作之前,该按钮为禁用状态,当机遇结果保存了新的文件后,该按钮为可用状态。此时点击该按钮,系统将弹出下拉框让你选择需要计算的工,点击后页面将立即开一个新的标签页,并将你保存的数据集一并带过去,在调整参数后即可提交新的任务。
4. 相关文献
[1] ResGen is a pocket-aware 3D molecular generation model based on parallel multiscale modelling. Zhang, O., et al. Nature Mach Intell (2023)
