Inno-Rescoring
1. Inno-Rescoring 概述
Inno-Rescoring 模块提供了一些基于机器学习算法的优秀打分函数。为了方便用户可以更直接的使用我们的重打分函数,Rescoring 模块支持用户在本地对接后上传相关的文件进行重打分,用户可以选择这些重打分函数重新评估蛋白质-配体结合姿势的结合亲和力。
2. 使用说明
用户只需要四个步骤就可以完成计算:上传预处理后蛋白-上传对接后配体-任务命名(可忽略)-提交任务(必点)。
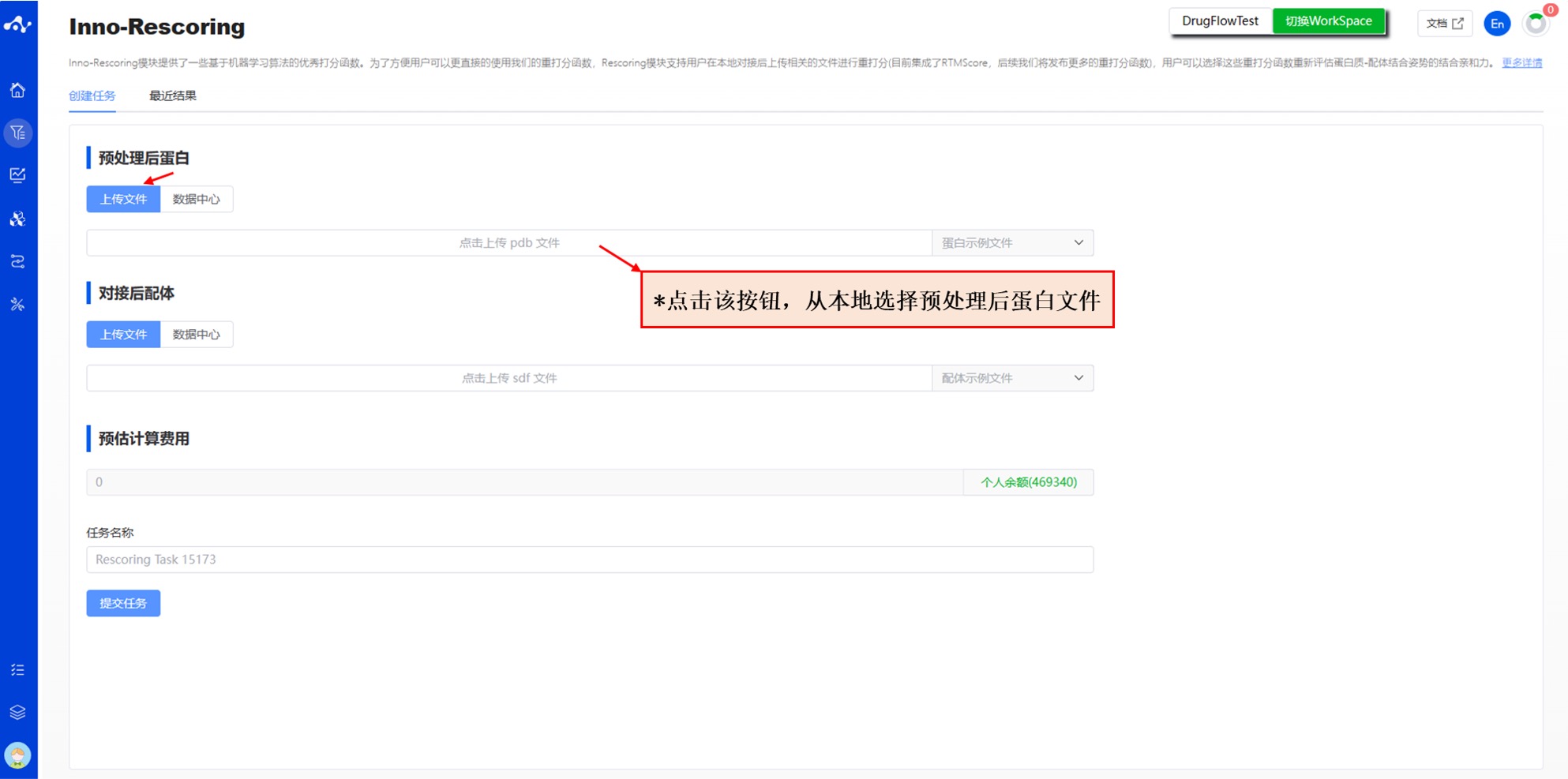
图 1. 上传预处理后蛋白和对接后配体——上传文件/数据中心
(1) 上传预处理后蛋白
平台提供了两种上传预处理蛋白的方式:上传文件和数据中心。
- 上传文件
复选框选中“上传文件”,通过点击下方按钮选择本地文件即可。选择完文件以后,该文件名称将显示在按钮上。
- 当前支持的文件格式: .pdb
- 数据中心
复选框选中“数据中心”,通过点击下方按钮页面出现弹窗,点击文件名称来选择数据中心的数据,点击完之后弹窗消失。
(2) 上传对接后配体
平台提供了两种上传对接后配体的方式:上传文件和数据中心。
- 上传文件
复选框选中“上传文件”,通过点击下方按钮选择本地文件即可。选择完文件以后,该文件名称将显示在按钮上。
- 当前支持的文件格式: .sdf
- 数据中心
复选框选中“数据中心”,通过点击下方按钮页面出现弹窗,点击文件名称来选择数据中心的数据,点击完之后弹窗消失。
在确定好所有的参数后,命名任务名称,点击提交即完成任务提交操作。
(3)运行进度和结果查看
提交任务后,页面会自动跳入当前页面的“最近结果”子页面中,您可以在该页面查看当前模块的任务运行状态(进度条),也可在右上角的“运行中“下拉框中查看所有模块正在运行的任务。当数据量较大时,系统会分批计算,因此只要有一批数据算完后(整个任务还在运行中),即可点击“结果详情”按钮进入结果页面,查看当前已完成计算的预测结果列表(未完成计算的分子暂不显示),并且可以在当前页面通过刷新来获取最新算完的数据。
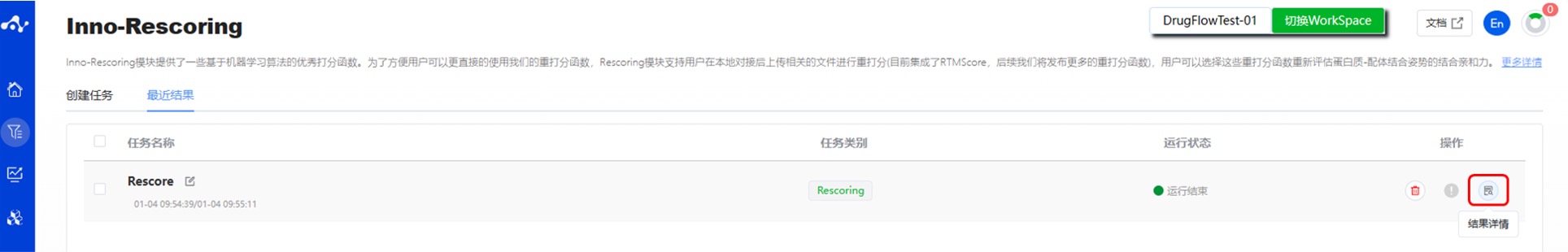
图 2. 查看结果
3. 结果分析
结果页面由顶部的筛选区、Summary 和中部的结果详情区组成。默认状态下结果详情区展示的是结果列表(还可以切换卡片),此时您可以查看所有得分,并对这些得分进行排序。
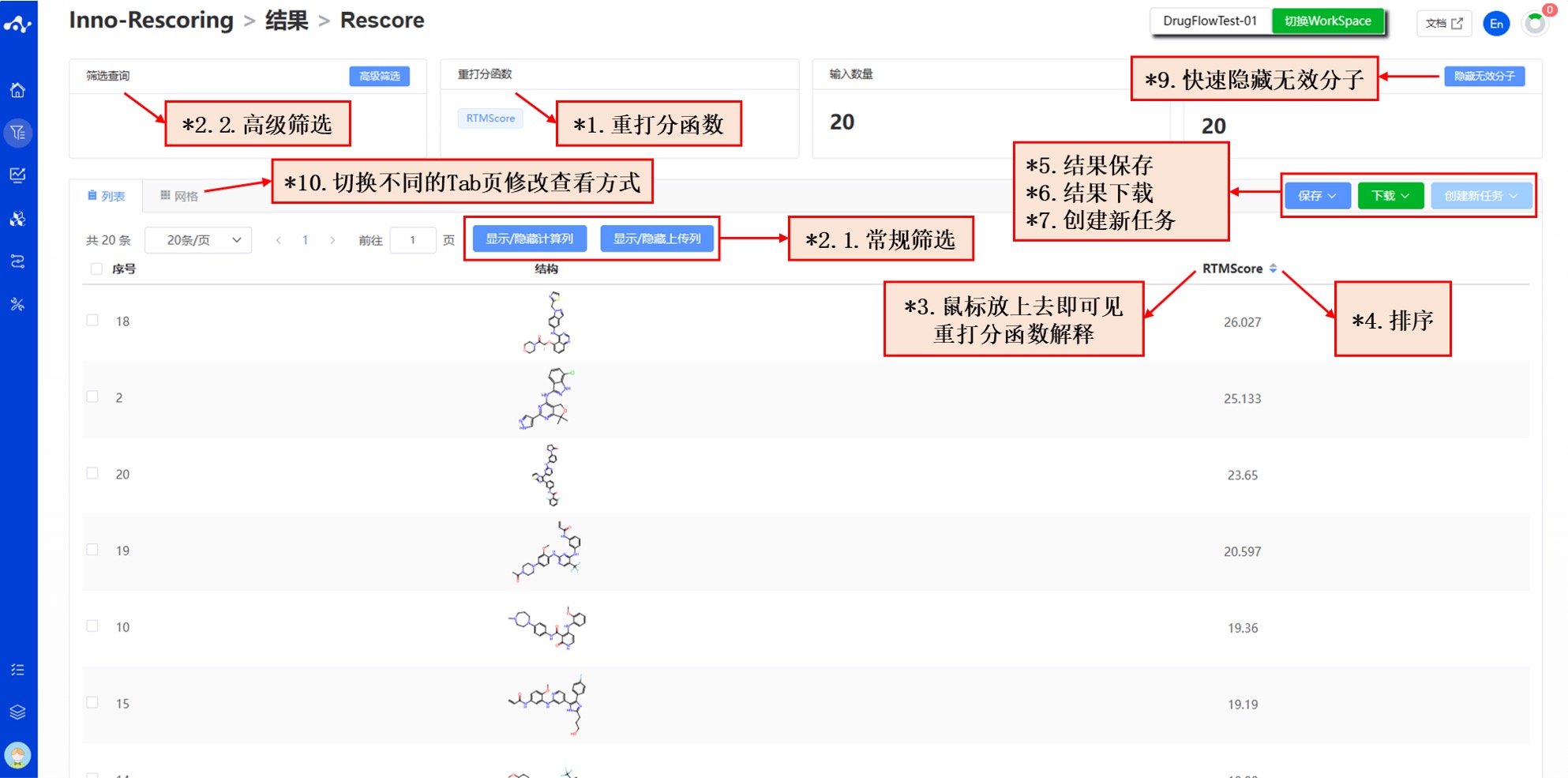
图 3. 结果页面功能分布
(1) 重打分函数
重打分函数为 RTMScore。
(2) 筛选
平台提供了常规筛选和高级筛选以满足用户的使用需求。
- 常规筛选
常规筛选可以显示/隐藏重打分函数。默认的结果列表展示计算的所有重打分函数,此时左边控制栏中为全选的状态。当您不想显示该重打分函数时,取消该重打分函数的选中即可,左边的结果列表将根据控制栏中的选择进行实时显示。在顶部还提供了两个快捷键“全选”和“不选”,方便用户快速选择。
- 高级筛选
在常规筛选的基础上,高级筛选提供了范围筛选,可进一步筛选出得分值指定范围内的分子,以排除不符合预期结果的分子。
(3) 重打分函数解释
把鼠标移入每个重打分函数的名称上,可查看该重打分函数对应的解释。
(4) 排序
点击结果列表中的重打分函数名字可重新排序,如 RTMScore,点击一次为升序,再点一次为降序,再点一次即恢复原始排序。
(5) 保存
点击“保存”,系统将弹出下拉框让您选择保存的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据您的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至数据中心。
(6) 下载
点击“下载”,系统将弹出下拉框让您选择下载的文件格式(目前仅支持.csv/.sdf)。确定好下载的文件样式后,系统将根据您的显示隐藏列的条件和高级筛选的条件,保存相应的数据为 sdf 或 csv 文件至您的本地设备上。
(7) 创建新任务
创建新任务的前提是先保存数据为一个文件,在未执行保存操作之前,该按钮为禁用状态,当基于结果保存了新的文件后,该按钮为可用状态。此时点击该按钮,系统将弹出下拉框让您选择需要计算的新任务种类,点击后页面将立即开一个新的标签页,并将您保存的数据集一并带过去,在调整参数后即可提交新的任务。
(8) 隐藏无效分子
针对 SMILES 错误或者后台无法解析的分子,算法无法进行正确的计算,在这种情况下,任务不受影响,但分子被定义为无效分子。用户可以通过“隐藏无效分子”的按钮,快速过滤掉该部分的分子。
(9)卡片页面
在卡片页面,我们提供了一个更加简洁的方式查看结果,您能看到该分子的结构及重打分函数 RTMScore 分值。
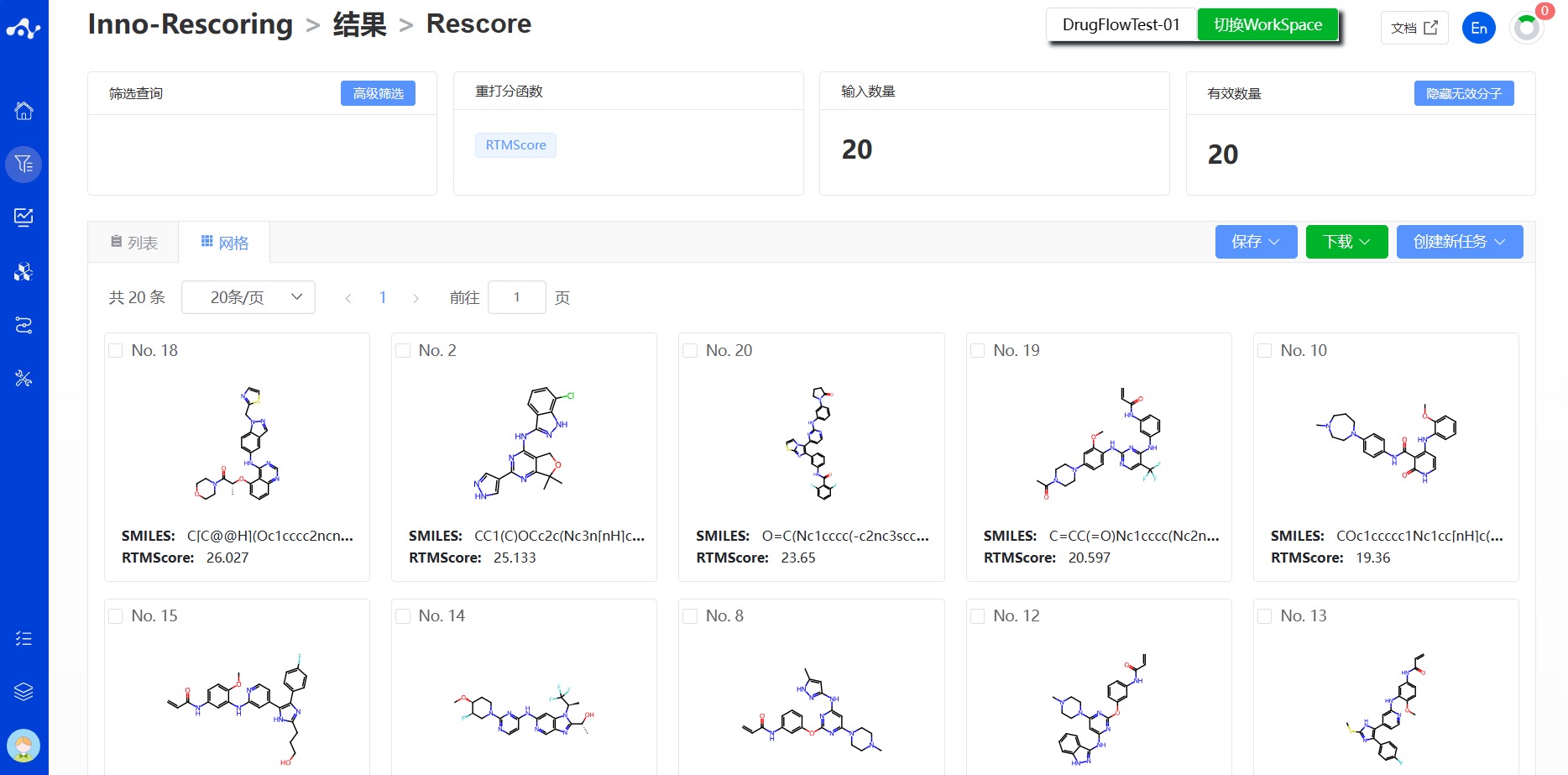
图 4. 结果的网格页面
4. 相关算法介绍
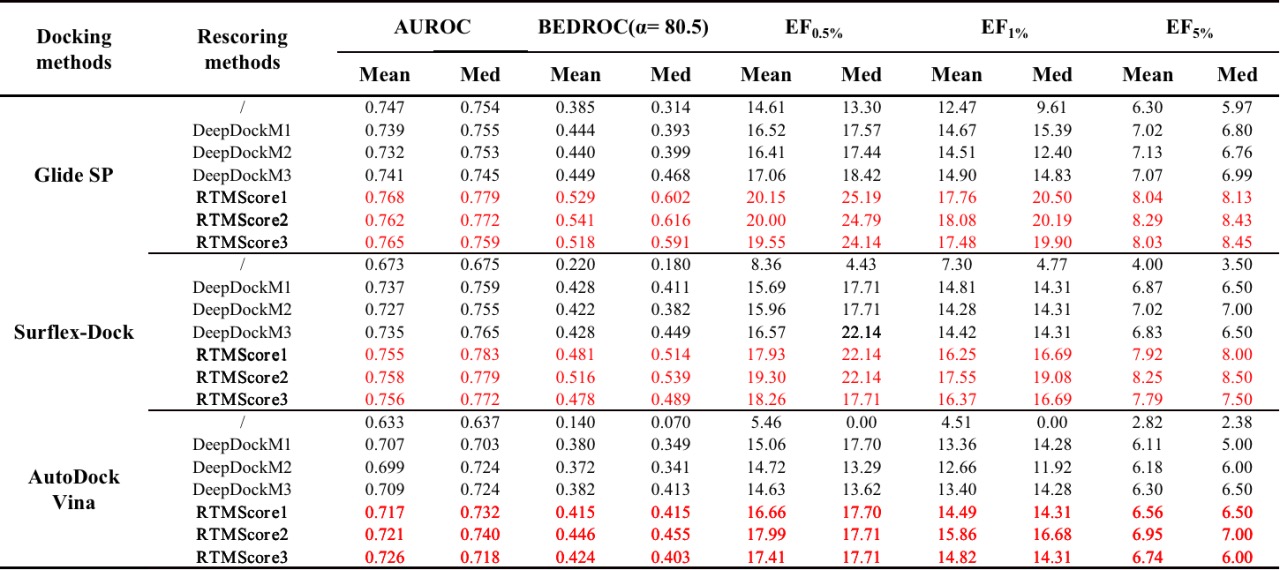
RTMScore 是一种新型的蛋白质-配体结合亲和度预测评分函数,我们通过 Graph Transformer 提取蛋白质的氨基酸残基以及配体的原子节点特征,并通过混合密度网络(Mixture Density Network, MDN)获取蛋白质各氨基酸残基和配体各原子间距离的概率密度分布,最后将其转化为统计势以用于蛋白-配体间结合强度的评估。结果表明,RTMScore 的对接能力和筛选能力在 CASF-2016 标准数据集上显著超越了当前的其他主流方法,其在有无天然构象的存在下分别可取得 97.3%和 93.4%的平均 top1 对接成功率(DeepDock 和 PIGNet 在无天然构象时仅为 87.0%);而在筛选能力评估中可取得 66.7%的平均 top1 成功率和 28.00 的 1%富集因子(DeepDock 和 PIGNet 的两项指标分别为 55.4%、19.60 以及 43.9%、16.41)。同时我们进一步在 DEKOIS2.0 数据集上评估了 RTMScore 筛选能力,结果表明 RTMScore 的富集能力明显优于以相似策略构建所得的 DeepDock 和经典方法 Glide SP,详见参考文献[1]。
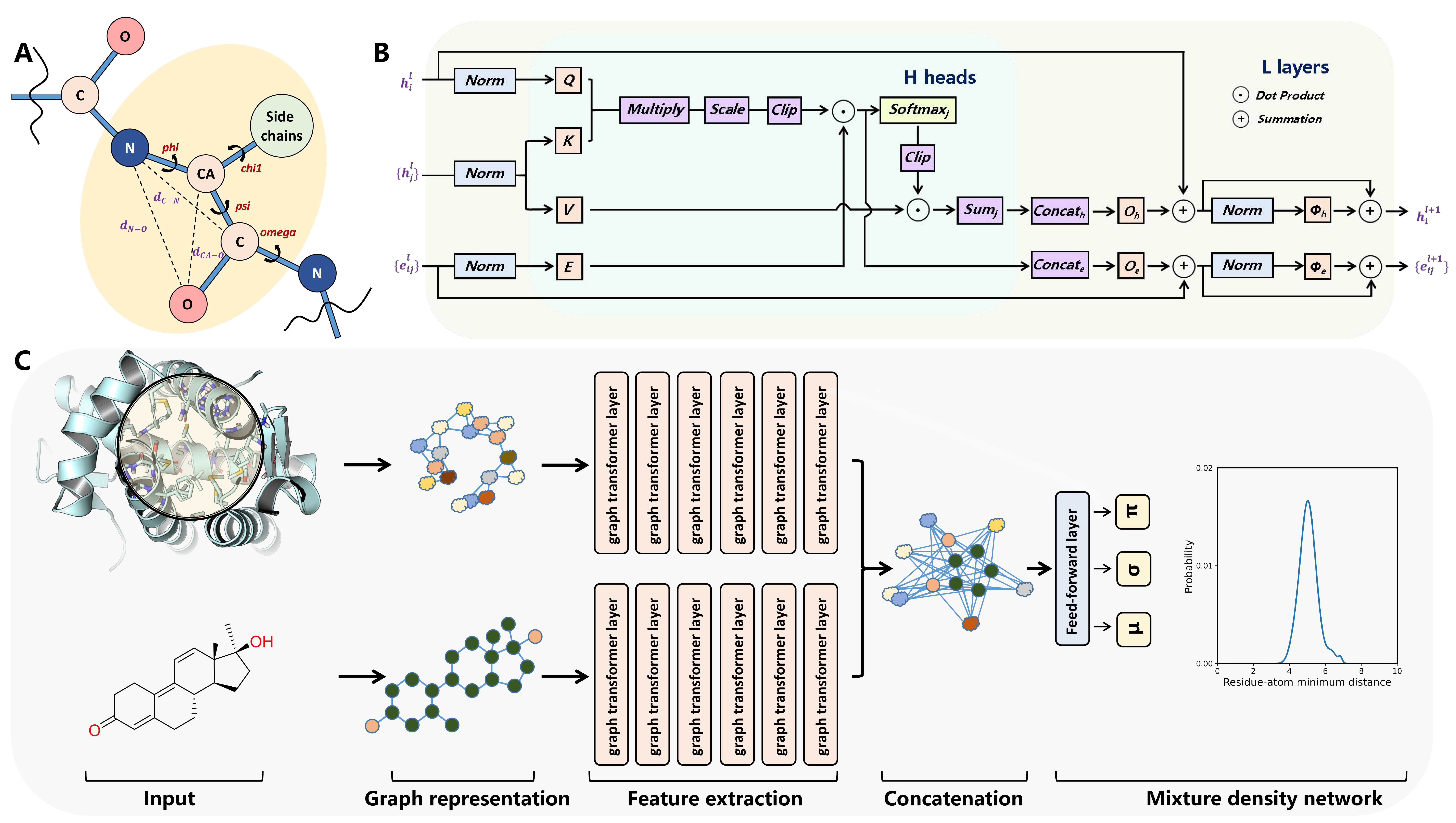
图 5. RTMScore 模型框架
表 1. RTMScore 与其他先进方法在 CASF-2016 基准测试上的对接能力和筛选能力的比较。
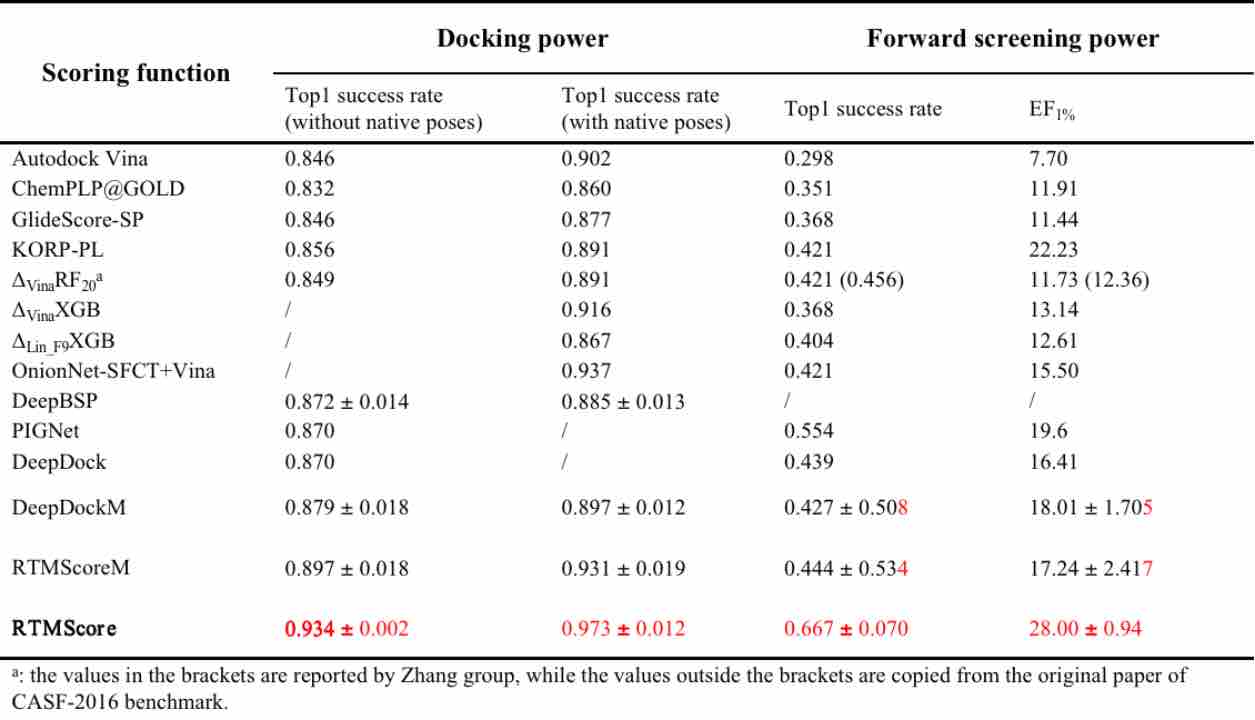
表 2. RTMScore 在 DEKOIS2.0 数据集上的筛选能力评估结果
5. 相关文献
[1] Boosting Protein-Ligand Binding Pose Prediction and Virtual Screening Based on Residue−Atom Distance Likelihood Potential and Graph Transformer. Shen C, Zhang X, Deng Y, et al. J Med Chem. 2022. doi:10.1021/acs.jmedchem.2c00991
