Inno-QSAR
1. Inno-QSAR 概述
定量结构-活性关系(Quantitative Structure-activity Relationship, QSAR)是一种研究化合物的化学结构、物理化学性质与其生物活性之间的关系,从而建立定量(定性)的预测模型的方法。随着计算机技术的迅速发展,现在已经发展成基于各种机器学习(Machine learning, ML)算法的 QSAR 模型,随机森林(Random Forest, RF)、极限梯度提升(eXtreme Gradient Boosting, XGBoost)等 ML、DL 算法已逐步替代了最开始的 Hansch 方法和 Free-wilson 方法。通过使用这些算法来学习一系列已知生物活性的化合物及其相应的结构或物理性质之间的关系,可以建立一个 QSAR 分类或回归模型,从而有效的区分活性分子和非活性分子或预测新化合物的生物活性。
对于一个模型而言,最关键的是模型的性能,而影响模型性能的主要因素有 3 个,分别为数据集、描述符和算法。
数据集。数据集是一个模型的基石,它直接决定了最终模型的可靠性和实用性。一个结构越多样,数据越多的数据集,意味着模型的应用阈越广,实用性越强。因此用户在建模之前,应该尽可能的收集到更多的数据。
描述符(可选项,仅适用于 ML 算法)。描述符是一种更为复杂的分子表征,它使用特定的数值来表示某一个特征,而不是简单的二进制编码(分子指纹)。在建模时,用户应该根据其研究目的来选择合适的描述符来表征分子相关信息。
算法。基于 ML/DL 的算法会比传统的建模方法有明显优势,但同时它也存在一定的缺陷,即参数多并且难调,需要多次调参才能构建一个性能良好的模型。为此我们简化了算法调参的过程,仅暴露了一些关键的参数,并提供了默认的参数数据,使得用户可以快速入手并建立效果优秀的 QSAR 模型。
Inno-QSAR 模块提供了基于自有数据进行快速 AI 建模的能力,支持主流数据清洗、数据批分、描述符计算以及 ML/DL 算法选择。用户可以选择自动化建模模式或自主调整参数模式,以便获得最佳模型。结果页面还提供了完整的模型评估及统计图表展示功能,所建模型可方便地集成到系统流程中。
2. 使用说明
用户只需要六个步骤就可以完成计算:选择模型类型-上传数据-设置建模数据-设置建模方法-任务命名(可忽略)-提交任务(必点)。
(1) 选择模型类型
用户根据自身需求选择构建模型的种类:分类模型和回归模型。
(2) 上传数据
上传数据包括 Training Set 和 Test Set。
平台提供了两种数据输入方式:上传文件和数据中心。
- 上传文件
复选框选中“上传文件”,通过点击下方按钮选择本地文件即可。选择完文件以后,该文件名称将显示在按钮上,右边将显示文件内容。
- 当前支持的文件格式: .sdf/.csv;如上传 csv 文件,则需指定 smiles 列。如上传 sdf 文件,则无需指定 smiles 列。
- 数据中心
复选框选中“数据中心”,通过点击下方按钮页面出现弹窗,点击文件名称来选择数据中心的数据,点击完之后弹窗消失。
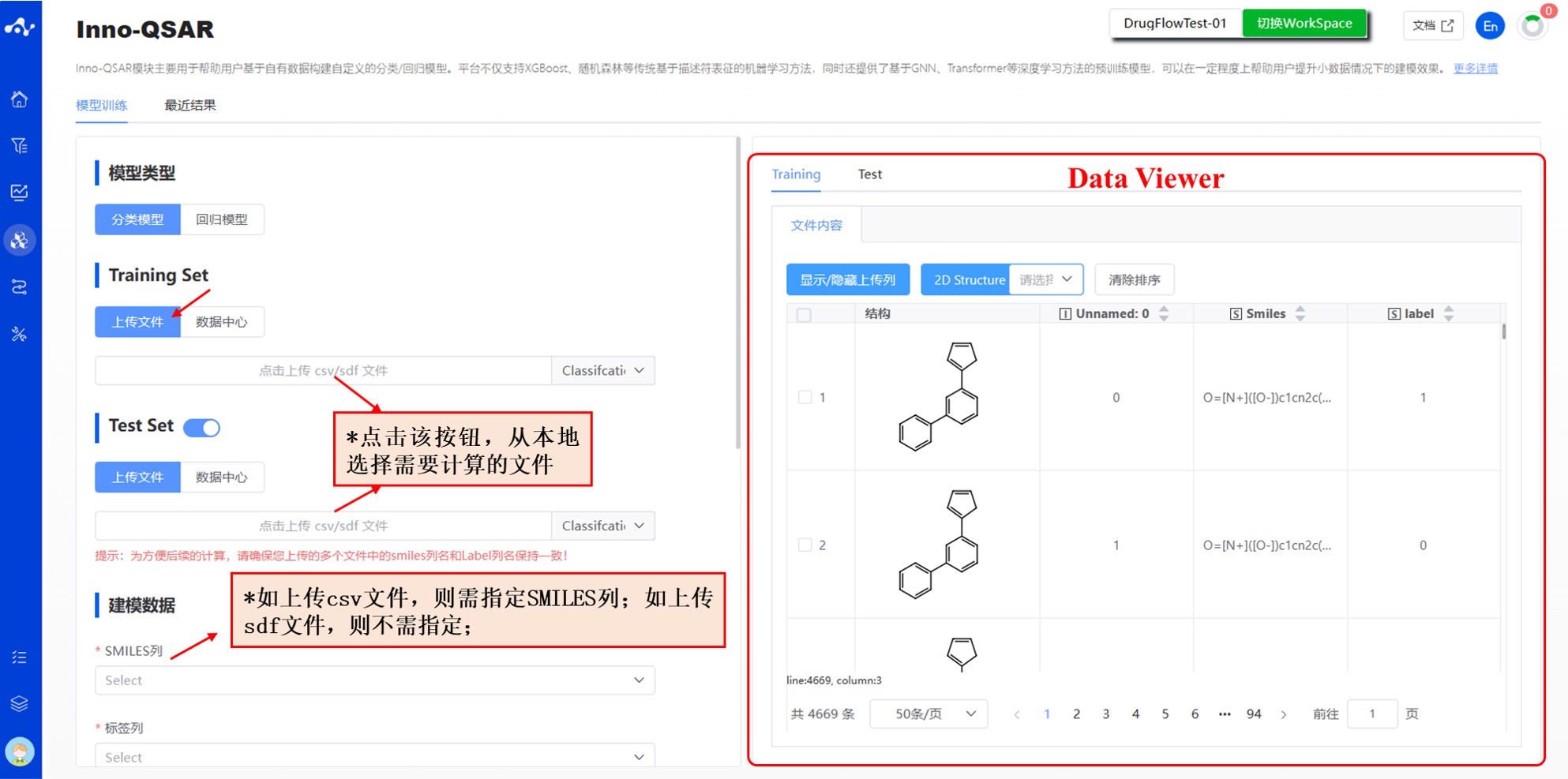
图 1. 输入方式——上传文件/数据中心
(3) 设置建模数据
如上传了 csv 文件,则需指定 smiles 列。
指定标签列。
如果是分类模型,则需指定正例值。
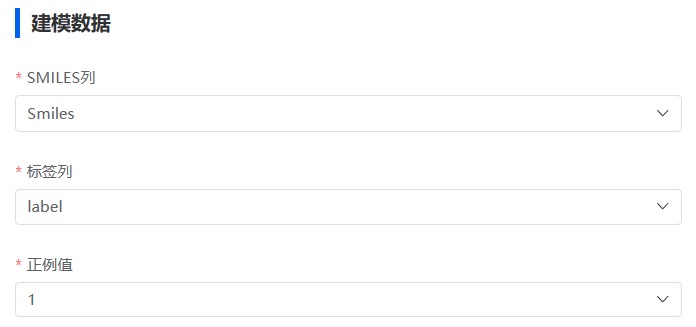
图 2. 设置建模数据
(4) 设置建模方法
平台提供两大类建模方法可供选择,包括基于描述符和基于结构。基于描述符的算法有:AutoML、XGBoost 和 RF;基于结构的算法有:Transformer(Pretrain)和 GNN(Pretrain)。
- 基于描述符
基于描述符的方法针对的是机器学习方法,首先您需要从 AutoML、RF 和 XGBoost 中选择一种算法。进一步地,设置描述符,平台提供了两种设置描述符的方式,包括 Compute New Descriptor 和 User-Defined。
- Compute New Descriptor
用户从 ECFP4、ECFP6、MACCS 和 MoleculeDescriptors 中选择一种或多种描述符,平台基于用户指定的描述符种类自动计算。
ECFP4/6. ECFP(Extended-Connectivity Fingerprints)是一种圆形拓扑指纹,包含高度特定的原子信息,可用于表示子结构特征。ECFP 后面的数字表示圆的最大直径,它指定了每个原子要考虑的最大圆邻域。默认直径为 4,可以满足相似性搜索和聚类的要求,直径为 6/8 有利于较大的结构片段,更适合活性预测。
MACCS. MACCS 描述符是一种结构描述符,包含 166 个最常见的子结构特征。
Molecule Discriptors. Molecule Discriptors 指的是 RDKit 中的 115 个 2D 描述符。
- User-Defined Descriptor
用户提前计算好描述符,并将其保存在上传文件中,在此处指定描述符所在列。
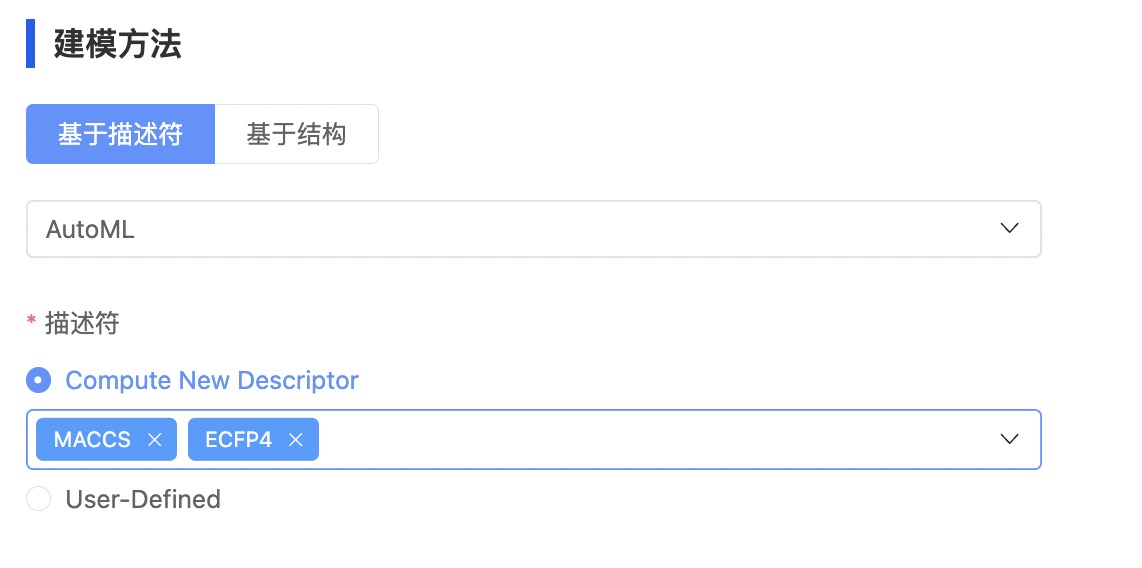
图 3. 设置建模方法——计算描述符
- 基于结构
基于结构的方法主要针对的是深度学习方法,首先您需要从 Transformer(Pretrain) 和 GNN(Pretrain)中选择一种算法,我们预先对这两种算法在大规模数据上进行了预训练。
进一步地,用户依次对 num_train_epochs、batch_size 和 learning_rate 进行设置。

图 4. 设置建模方法——基于结构
在确定好所有的参数后,命名任务名称,点击提交即完成任务提交操作。
(5) 运行进度和结果查看
提交任务后,页面会自动跳入当前页面的“最近结果”子页面中,您可以在该页面查看当前模块的任务运行状态(进度条),也可在右上角的“运行中“下拉框中查看所有模块正在运行的任务。待任务结束计算后,即可点击“结果详情”按钮进入结果页面,查看当前已完成计算的结果。
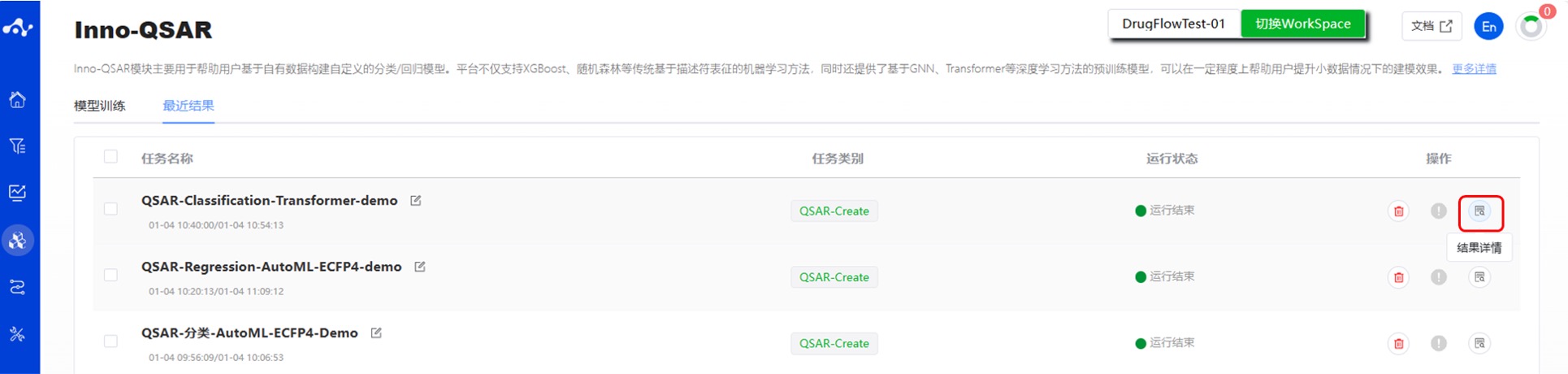
图 5. 查看结果
3. 结果分析
结果页面由 3 个部分组成,分别为数据分析、骨架分析和结果详情。
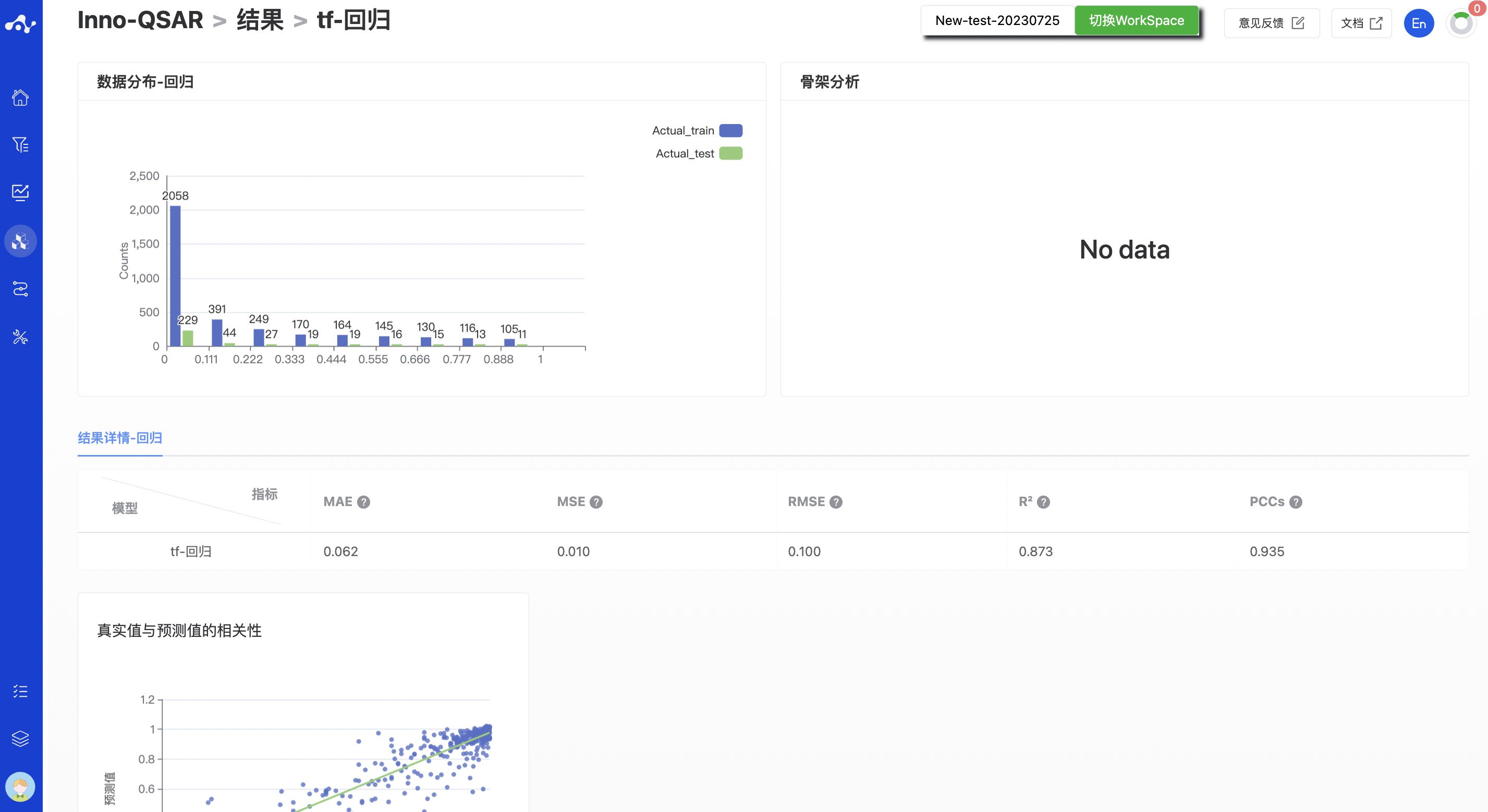
图 6. Inno-QSAR 结果页面布局
(1) 数据分析
针对预处理之后的训练集和测试集数据,绘制其统计分布图,以协助用户快速了解实际进入模型的数据分布。
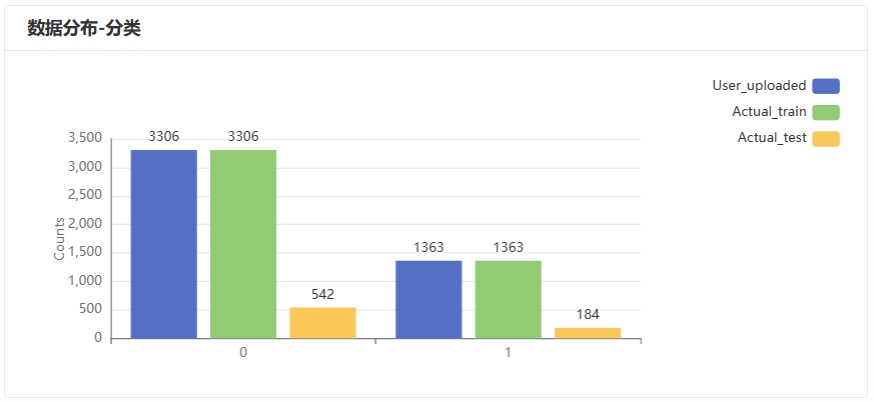
图 6.数据分布柱状图-分类
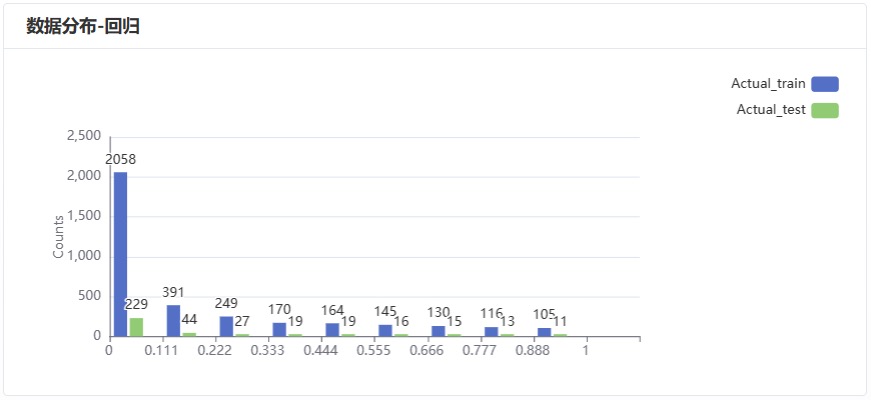
图 7.数据分布柱状图-回归
(2) 骨架分析
当前平台暂时不提供骨架分析,后续将会对其进行完善。
平台对训练集、测试集的数据,根据 Murcko 骨架做一个骨架分析,并在页面上显示排名前 10 的骨架结构。
回归模型的骨架分类:训练集、测试集;
分类模型的骨架分类:1-训练集,1-测试集,0-测试集,0-训练集。
(3) 结果详情
- 分类模型
根据分类模型的特点,基于测试集计算了相应的评估指标,包括准确率,ROC-AUC、PRC-AUC、F1-Score、精确率和召回率,并提供了 ROC 曲线和 PR 曲线。
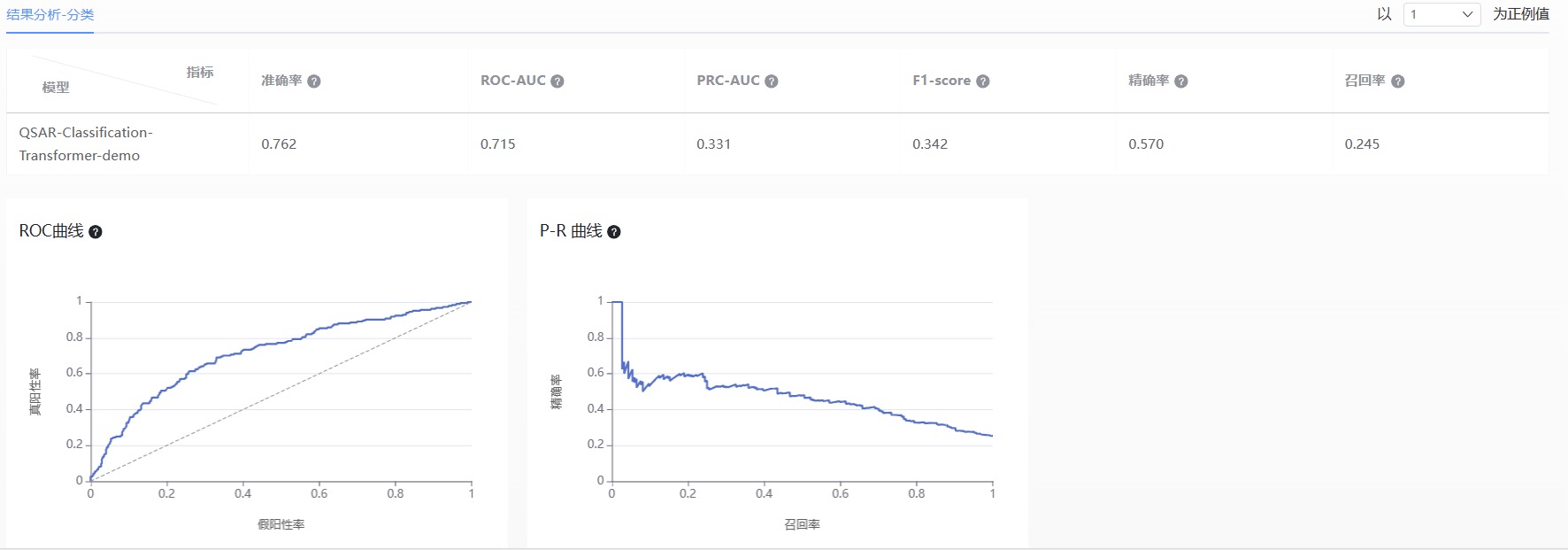
图 8.分类模型结果分析图
- 回归模型
根据回归模型的特点,基于测试集计算了相应的评估指标,包括 MAE、MSE、RMSE、R2 和 PCCs,并提供了真实值和预测值之间相关性的散点图。
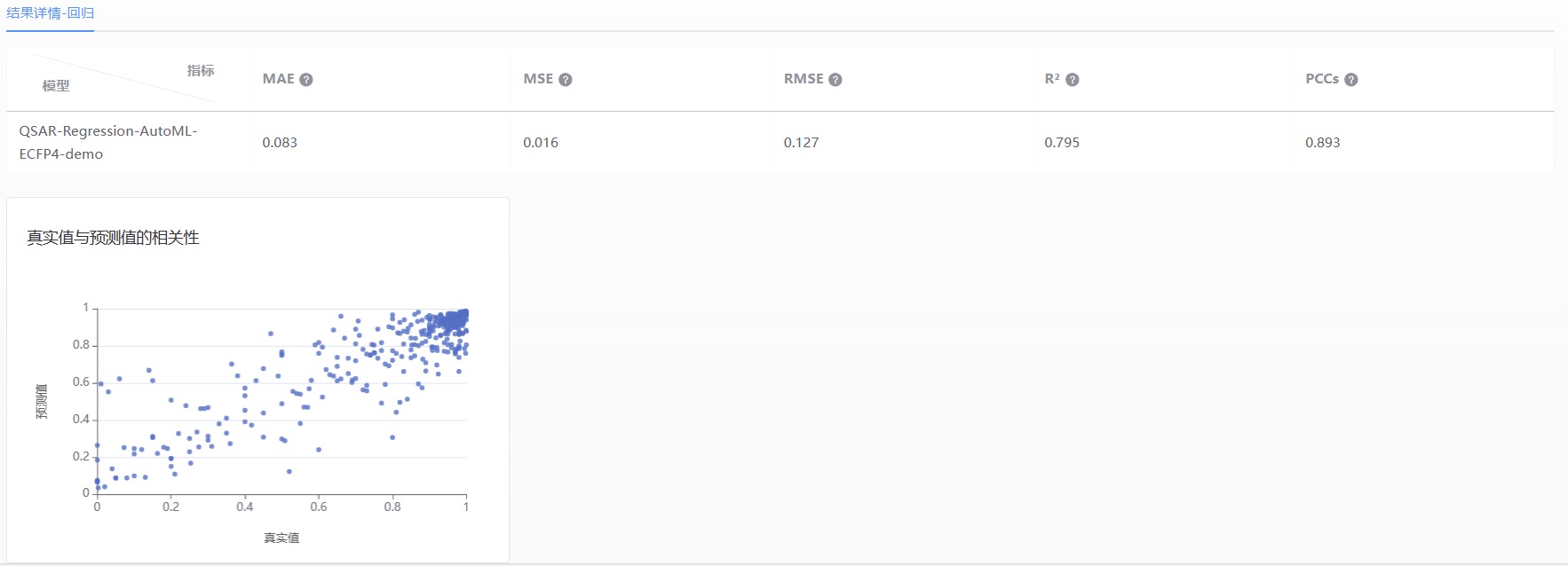
图 9. 回归模型结果分析图
4. 相关算法介绍
(1) AutoML
AutoML 中包含 11 种算法,基于用户上传的数据,通过一些自动化处理(包括处理缺失数据、手动特征转换、数据分割、模型选择、算法选择、超参数选择和调整、集合多个模型)替换传统耗时的手动处理步骤,并应用 L-layer stacking & n-repeated k-bagging 的 ensembling 技术,在提升计算速度的同时也极大的提高了模型的准确性。AutoML 会根据输入的数据自适应地剔除部分不适用的模型,再基于构建的多个模型进行加权,最终 AutoML 输出的结果是运行成功的算法模型的加权平均值。
上述提及的 11 种算法分别为:
Random Forest
Extremely Randomized Trees
K-Nearest Neighbors
LightGBM Boosted Trees
CatBoost Boosted Trees
XGBoost Model
Linear Regression
PyTorch neural network models
VowpalWabbitModel
Fastai v1 neural network models
AutoGluon Deep Neural Networks
(2) XGBoost
XGBoost 是一种基于集成学习的机器学习算法,主要是通过梯度提升(Gradient Boosting)来构建一个集成模型。XGBoost 通过多次迭代,将弱分类器进行叠加,使得每次迭代都可以更好地拟合残差。同时,XGBoost 还利用了正则化来控制模型的复杂度,避免过拟合。在模型训练过程中,平台应用网格搜索+5 折交叉验证的组合方法,来确定最优参数,以此来提高模型的准确性和泛化能力,避免由于数据分布不均等问题导致模型性能偏差。
(3) RF(Random Forest)
RF 是一种基于决策树的机器学习算法,它通过对数据集进行有放回抽样(Bootstrap 抽样),并对每个样本进行多次随机选择特征,来构建多个决策树。最后,将这些决策树进行结合(投票或平均)来预测新的数据。RF 可以有效地处理高维数据和非线性关系,同时也可以减轻过拟合的风险。在模型训练过程中,平台应用网格搜索+5 折交叉验证的组合方法,来确定最优参数,以此来提高模型的准确性和泛化能力,避免由于数据分布不均等问题导致模型性能偏差。
(4) Transformer(Pretrain)
Transformer(Pretrain)是碳硅智慧自主研发的一个预训练模型。基于 330 万条小分子数据,以分子的 1D、2D 和 3D 信息作为输入,采用 “模态先对齐,后融合” 的思想,引入 Contrast Loss、Matching Loss 和 Momentum Encoder 策略对分子 global 特征进行约束,homo-lumo-gap 作为监督信号约束分子的物理性质,使用 3D 分子构象去噪任务约束分子的 3D 结构,最终应用分子片段定位的代理任务对分子的局部信息进行约束。通过 MOE(Mixture-of-Experts)结构极大的增强了模型多模态信息的适配能力。经多个数据集的验证,结果表明该预训练模型可以明显的提高小数据集的建模效果。
(5) GNN(Pretrain)
GNN(Pretrain)是碳硅智慧自主研发的一个预训练模型,以原子特征和键特征编码的分子图(包括 9 种原子特征和 5 种键特征用于表征原子、化学键及其局部环境)作为输入,通过共享参数的多个个 RGCN 层从子结构中提取通用特征,并应用注意力层为不同的子结构分配不同的注意力权重,训练了一个性能良好的预训练模型。其优势在于:1)正则化效应,模型支持使用相同数量的参数学习更多任务;2)迁移学习效应,ADMET 数据具有相关性,而相关任务共享部分隐藏层参数有助于提取通用特征;3)数据增强效应,ADMET 数据稀疏,模型同时对所有的性质进行训练,避免在小任务上出现过拟合。
4. 相关文献
暂无
